Difference between revisions of "Dynamico-giant"
(Created page with "[transferred from github, now active here] # 1. Introduction "Welcome to the dynamico-giant wiki" Dynamico-giant est un dépôt git qui permet d'installer le modèle Dynami...") |
m |
||
| (6 intermediate revisions by 3 users not shown) | |||
| Line 1: | Line 1: | ||
[transferred from github, now active here] | [transferred from github, now active here] | ||
| − | + | = 1. Introduction = | |
| − | + | "Welcome to the dynamico-giant wiki" | |
Dynamico-giant est un dépôt git qui permet d'installer le modèle Dynamico avec les fichiers de configs propres aux planètes géantes gazeuses et géantes glacées. Ci-dessous, vous trouverez presque (car nous ne sommes pas parfaits) toutes les informations sur l'installation, la compilation du modèle et les paramètres que vous pouvez modifier pour faire tourner le modèle. | Dynamico-giant est un dépôt git qui permet d'installer le modèle Dynamico avec les fichiers de configs propres aux planètes géantes gazeuses et géantes glacées. Ci-dessous, vous trouverez presque (car nous ne sommes pas parfaits) toutes les informations sur l'installation, la compilation du modèle et les paramètres que vous pouvez modifier pour faire tourner le modèle. | ||
| − | + | "Allons-y!" | |
| − | + | = 2. Installation = | |
| − | + | == A) Fichiers d'architecture == | |
| − | + | === - Modules === | |
Before installation, set environment (do it once) in your .bash_profile. | Before installation, set environment (do it once) in your .bash_profile. | ||
Modules used on Occigen: | Modules used on Occigen: | ||
| − | + | ||
| − | ulimit -s unlimited | + | <pre>ulimit -s unlimited |
# modules | # modules | ||
module purge | module purge | ||
| Line 29: | Line 29: | ||
module load nco | module load nco | ||
module load qt | module load qt | ||
| − | module load python | + | module load python</pre> |
| − | + | Modules used on Ciclad: | |
| − | + | <pre>ulimit -s unlimited | |
| − | |||
| − | ulimit -s unlimited | ||
# modules | # modules | ||
module purge | module purge | ||
| Line 41: | Line 39: | ||
module load openmpi/1.6.5-ifort | module load openmpi/1.6.5-ifort | ||
module load hdf5/1.8.18-parallel-ifort | module load hdf5/1.8.18-parallel-ifort | ||
| − | module load netcdf4/4.4.1.1-parallel-ifort | + | module load netcdf4/4.4.1.1-parallel-ifort</pre> |
| − | |||
Beware, if you change version of model (newer or older version), it's possible that you have to change modules... | Beware, if you change version of model (newer or older version), it's possible that you have to change modules... | ||
| − | + | === - Installation sur un nouveau cluster === | |
En pratique LMDZ.COMMON, ICOSA_LMDZ et IOIPSL peuvent utiliser exactement le même fichier arch.fcm ; mais celui pour ICOSAGCM est légèrement différent (les %FPP_DEF diffèrent, peut-être aussi le %FPP). | En pratique LMDZ.COMMON, ICOSA_LMDZ et IOIPSL peuvent utiliser exactement le même fichier arch.fcm ; mais celui pour ICOSAGCM est légèrement différent (les %FPP_DEF diffèrent, peut-être aussi le %FPP). | ||
| − | make_icosa_lmdz doit être lancé avec -arch_path ../ARCH puisque les arch.env et arch.path communs se trouvent dans ../ARCH (l'option -arch_path ne concerne d'ailleurs que les fichiers arch.env et arch.path; le fichier arch.fcm recherché sera toujours celui dans le | + | make_icosa_lmdz doit être lancé avec -arch_path ../ARCH puisque les arch.env et arch.path communs se trouvent dans ../ARCH (l'option -arch_path ne concerne d'ailleurs que les fichiers arch.env et arch.path; le fichier arch.fcm recherché sera toujours celui dans le "arch" de chacun des modèles. Donc il faut bien mettre pour chacun des quatre modèles (LMDZ.COMMON, ICOSA_LMDZ,IOIPSL et ICOSAGCM) le arch.fcm dans le sous-dossier arch/ du modèle correspondant. |
L'installation d'IOIPSL doit se faire à partir du bon script bash. Si vous utilisez un server autre qu'occigen, il vous faut changer le nom du script à utiliser dans install_ioipsl.sh. Par exemple, il vous faudra utiliser install_ioipsl_ciclad-ifort.bash pour CICLAD. | L'installation d'IOIPSL doit se faire à partir du bon script bash. Si vous utilisez un server autre qu'occigen, il vous faut changer le nom du script à utiliser dans install_ioipsl.sh. Par exemple, il vous faudra utiliser install_ioipsl_ciclad-ifort.bash pour CICLAD. | ||
| − | XIOS est écrit en C++, et pas en Fortran. Le fichier arch.fcm correspondant est donc nécessairement différent de celui des quatre autres modèles. Pour créér ce arch.fcm, prendre exemple sur les fichiers .fcm déjà présent dans XIOS/arch/, avec une architecture similaire à celle du nouveau cluster. En particulier, il faut utiliser des versions de gcc/gfortran | + | XIOS est écrit en C++, et pas en Fortran. Le fichier arch.fcm correspondant est donc nécessairement différent de celui des quatre autres modèles. Pour créér ce arch.fcm, prendre exemple sur les fichiers .fcm déjà présent dans XIOS/arch/, avec une architecture similaire à celle du nouveau cluster. En particulier, il faut utiliser des versions de gcc/gfortran > 6+. Il faut absolument avoir une bibliothèque HDF5 compilée en parallèle, ainsi que netcdf-C et netcdf-fortran (et peut être aussi netcdf-Cxx) compilé en parallèle. Sans ça, il sera peut-être possible de compiler le modèle, mais pas de lancer une simulation en utilisant XIOS. |
| − | Il faut absolument avoir une bibliothèque HDF5 compilée en parallèle, ainsi que netcdf-C et netcdf-fortran (et peut être aussi netcdf-Cxx) compilé en parallèle. Sans ça, il sera peut-être possible de compiler le modèle, mais pas de lancer une simulation en utilisant XIOS. | ||
| − | + | == B. Download == | |
Download structure | Download structure | ||
| Line 63: | Line 59: | ||
On occigen: | On occigen: | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | <pre>cd $SCRATCHDIR | |
| + | git clone https://github.com/aymeric-spiga/dynamico-giant.git [optional different name]</pre> | ||
| + | == C. Install == | ||
Install code | Install code | ||
| − | + | ||
| − | cd dynamico-giant | + | <pre>cd dynamico-giant |
./install.sh | ./install.sh | ||
| − | ./install_ioipsl.sh | + | ./install_ioipsl.sh</pre> |
| − | |||
| − | |||
A login and a password are necessary to install IOIPSL (only for the first time, after that we can save them). Please contact TGCC computing center to get the login/password. | A login and a password are necessary to install IOIPSL (only for the first time, after that we can save them). Please contact TGCC computing center to get the login/password. | ||
| − | After that, please add | + | After that, please add "$PWD"/FCM_V1.2/bin/ to PATH environment variable. |
Ant that's all, we can now change parameters of files or compile the code to do a test (part 6) or eat a beautiful tartiflette (can be also do during compilation). | Ant that's all, we can now change parameters of files or compile the code to do a test (part 6) or eat a beautiful tartiflette (can be also do during compilation). | ||
| − | + | = 3. General parameters = | |
| − | + | ||
| + | == A) Mesh grid & resolution == | ||
On run_icosa.def: | On run_icosa.def: | ||
| − | + | ||
| − | nbp -- | + | <pre>nbp --> number of subdivision on a main triangle: integer (default=40) |
nbp = sqrt((nbr_lat x nbr_lon)/10) | nbp = sqrt((nbr_lat x nbr_lon)/10) | ||
nbp 20 40 80 160 | nbp 20 40 80 160 | ||
T-edge length (km) 500 250 120 60 | T-edge length (km) 500 250 120 60 | ||
| − | Example: nbp(128x96)=35 - | + | Example: nbp(128x96)=35 -> 40 |
| − | nbp(256x192)=70 - | + | nbp(256x192)=70 -> 80 |
| − | nbp(360x720)=160 - | + | nbp(360x720)=160 -> 160 |
| − | nsplit_i, nsplit_j -- | + | nsplit_i, nsplit_j --> sub splitting of main rhombus: integer |
Example: for nbp=80, nsplit_i=4,nsplit_j=6 | Example: for nbp=80, nsplit_i=4,nsplit_j=6 | ||
| − | nbp/nsplit_{i,j} = 20 | + | nbp/nsplit_{i,j} = 20 > 10 & 13 > 10 --> GOOD</pre> |
| − | + | === - Remapping === | |
| − | |||
| − | |||
Pour le remapping, il faut aller dans context_lmdz_physics.xml changer les paramètres ni_glo et nj_glo par exemple pour remapper sur du lat/lon à 360 pts en latitude et 720 pts en longitude (0.5°). | Pour le remapping, il faut aller dans context_lmdz_physics.xml changer les paramètres ni_glo et nj_glo par exemple pour remapper sur du lat/lon à 360 pts en latitude et 720 pts en longitude (0.5°). | ||
| − | + | <code><domain id="dom_regular" ni_glo="720" nj_glo="360" type="rectilinear"></code> | |
| − | + | == B) Time == | |
* ndays is number of days to simulate. | * ndays is number of days to simulate. | ||
* day_step is number of dynamical time step per day | * day_step is number of dynamical time step per day | ||
| − | * Dynamics called every day_length(in s) / day_step | + | * Dynamics called every day_length(in s) / day_step per day |
* 1 ts (physical timestep) = (day_length(in s)/day_step) x itau_physics | * 1 ts (physical timestep) = (day_length(in s)/day_step) x itau_physics | ||
| − | * Physics called (day_step / itau_physics) | + | * Physics called (day_step / itau_physics) per day |
| − | * Physics called day_length(in s) / ts | + | * Physics called day_length(in s) / ts per day |
| − | * Radiative called every Physics x iradia | + | * Radiative called every Physics x iradia physical timestep |
| − | * Radiative called every iradia/Physics | + | * Radiative called every iradia/Physics days |
| − | + | == C) Sponge layer == | |
| − | + | <pre>iflag_sponge=0 for no sponge (default) | |
| − | iflag_sponge=0 for no sponge (default) | ||
iflag_sponge=1 for sponge over 4 topmost layers | iflag_sponge=1 for sponge over 4 topmost layers | ||
iflag_sponge=2 for sponge from top to ~1% of top layer pressure | iflag_sponge=2 for sponge from top to ~1% of top layer pressure | ||
| − | mode_sponge=1 for u,v -- | + | mode_sponge=1 for u,v --> 0 |
| − | mode_sponge=2 for u,v -- | + | mode_sponge=2 for u,v --> zonal mean |
| − | mode_sponge=3 for u,v,h -- | + | mode_sponge=3 for u,v,h --> zonal mean |
| − | tau_sponge -- | + | tau_sponge --> damping frequency at last layer |
| − | -- | + | --> e-5 medium / e-4 strong yet reasonable / e-3 very strong</pre> |
| − | |||
| − | |||
Spiga et al (2020): Shaw and Shepherd (2007) showed that the inclusion of sponge-layer parameterizations that do not conserve angular momentum (which is the case for Rayleigh drag), or allow for momentum to escape to space, implies a sensitivity of the dynamical results (especially zonal wind speed) to the choice for model top or drag characteristic timescale, because of spurious downward influence when momentum conservation is violated. | Spiga et al (2020): Shaw and Shepherd (2007) showed that the inclusion of sponge-layer parameterizations that do not conserve angular momentum (which is the case for Rayleigh drag), or allow for momentum to escape to space, implies a sensitivity of the dynamical results (especially zonal wind speed) to the choice for model top or drag characteristic timescale, because of spurious downward influence when momentum conservation is violated. | ||
Il ne faut donc pas ajouter de sponge layer pour les planètes géantes (iflag_sponge = 0) | Il ne faut donc pas ajouter de sponge layer pour les planètes géantes (iflag_sponge = 0) | ||
| − | + | == D) Dissipation == | |
Spiga et al (2020): A subgrid-scale dissipation term is included in our Saturn DYNAMICO GCM to prevent the accumulation of energy at scales close to the grid resolution, caused by the GCM not resolving the turbulent scales at which this energy is dissipated. This hyperviscosity term is written in our Saturn DYNAMICO model as an iterated Laplacian term on a given variable. The three variables denoted are vorticity, divergence, and potential temperature, chosen to set horizontal dissipation on respectively the rotational component of the flow (e.g. Rossby waves), the divergent component of the flow (e.g. gravity waves), and the diabatic perturbations (e.g. coming from the physical packages). | Spiga et al (2020): A subgrid-scale dissipation term is included in our Saturn DYNAMICO GCM to prevent the accumulation of energy at scales close to the grid resolution, caused by the GCM not resolving the turbulent scales at which this energy is dissipated. This hyperviscosity term is written in our Saturn DYNAMICO model as an iterated Laplacian term on a given variable. The three variables denoted are vorticity, divergence, and potential temperature, chosen to set horizontal dissipation on respectively the rotational component of the flow (e.g. Rossby waves), the divergent component of the flow (e.g. gravity waves), and the diabatic perturbations (e.g. coming from the physical packages). | ||
| − | + | <pre>tau_graddiv --> dissipation timescale of smallest wvl: u,v (gradiv) : real (default=5000) | |
| − | tau_graddiv -- | + | tau_gradrot --> dissipation timescale of smallest wvl: u,v (nxgradrot) : real (default=5000) |
| − | tau_gradrot -- | + | tau_divgrad --> dissipation timescale of smallest wvl: h (divgrad) : real (default=5000) |
| − | tau_divgrad -- | + | nitergdiv --> number of iterations for gradiv operator : integer (default=1) |
| − | nitergdiv -- | + | nitergrot --> number of iterations for nxgradrot operator : integer (default=1) |
| − | nitergrot -- | + | niterdivgrad --> number of iterations for divgrad operator : integer (default=1)</pre> |
| − | niterdivgrad -- | + | tau_graddiv,tau_gradrot,tau_divgrad correspondent au temps de dissipation (plus c'est petit, plus la dissipation est efficace). nitergdiv,nitergrot,niterdivgrad correspondent à l'ordre du laplacien (plus c'est grand, plus on dissipe sélectivement les petites échelles). |
| − | |||
| − | + | Attention, Trop dissiper -> instabilité numérique | |
| − | |||
| − | + | Pas assez dissiper -> instabilité dynamique trop forte | |
| − | |||
| − | Pas assez dissiper - | ||
L'ordre du laplacien dans les fichiers .def est suffisant. S'il faut changer la dissipation, il vaut mieux changer les temps de dissipation que l'ordre du laplacien (risque d'instabilité numérique). | L'ordre du laplacien dans les fichiers .def est suffisant. S'il faut changer la dissipation, il vaut mieux changer les temps de dissipation que l'ordre du laplacien (risque d'instabilité numérique). | ||
| − | + | === - Facteurs de dissipation === | |
A partir de ces valeurs de temps de dissipation, il est également possible d'ajouter 2 facteurs de dissipation (fac_mid et fac_up) qui permettent d'augmenter la dissipation à partir de certains niveaux. Le fac_mid permet de diminuer le temps de dissipation d'un facteur sur toute l'atmosphère jusqu'au bottom. Il y a une zone transition de quelques niveaux entre le bottom (le temp de dissipation au bottom est sans facteur) et quelques niveaux au-dessus où la valeur du temps de dissipation est avec ce facteur jusqu'au top. On ne peut donc pas choisir l'altitude et la zone de transition avec ce facteur. Quant au fac_up, il permet de diminuer le temps de dissipation d'un facteur à partir d'une altitude et d'une épaisseur de zone de transition qu'on peut choisir. On peut utiliser la combinaison des 2 ou l'un des 2. Pour ne pas prendre en compte ces facteurs de dissipation, il faut qu'il soit mis à 1. | A partir de ces valeurs de temps de dissipation, il est également possible d'ajouter 2 facteurs de dissipation (fac_mid et fac_up) qui permettent d'augmenter la dissipation à partir de certains niveaux. Le fac_mid permet de diminuer le temps de dissipation d'un facteur sur toute l'atmosphère jusqu'au bottom. Il y a une zone transition de quelques niveaux entre le bottom (le temp de dissipation au bottom est sans facteur) et quelques niveaux au-dessus où la valeur du temps de dissipation est avec ce facteur jusqu'au top. On ne peut donc pas choisir l'altitude et la zone de transition avec ce facteur. Quant au fac_up, il permet de diminuer le temps de dissipation d'un facteur à partir d'une altitude et d'une épaisseur de zone de transition qu'on peut choisir. On peut utiliser la combinaison des 2 ou l'un des 2. Pour ne pas prendre en compte ces facteurs de dissipation, il faut qu'il soit mis à 1. | ||
| Line 164: | Line 148: | ||
Il est vivement conseillé de tracer votre profil de dissipation à partir des équations (dans vert_prof_dissip_icosa_lmdz.f90) afin d'être certain de leur allure. | Il est vivement conseillé de tracer votre profil de dissipation à partir des équations (dans vert_prof_dissip_icosa_lmdz.f90) afin d'être certain de leur allure. | ||
| − | + | == E) Rayleigh Friction == | |
| − | Spiga et al 2020: This drag plays the role devoted to surface friction on terrestrial planets, which allows to close the angular momentum budget through downward control (Haynes and McIntyre, 1987; Haynes et al., 1991). This could also be regarded as a zeroth-order parameterization for Magneto-HydroDynamic (MHD) drag as a result of Lorenz forces acting on jet streams putatively extending to the depths of Saturn's interior (Liu et al., 2008; Galanti et al., 2019), much deeper than the shallow GCM's model bottom. | + | Spiga et al 2020: This drag plays the role devoted to surface friction on terrestrial planets, which allows to close the angular momentum budget through downward control (Haynes and McIntyre, 1987; Haynes et al., 1991). This could also be regarded as a zeroth-order parameterization for Magneto-HydroDynamic (MHD) drag as a result of Lorenz forces acting on jet streams putatively extending to the depths of Saturn's interior (Liu et al., 2008; Galanti et al., 2019), much deeper than the shallow GCM's model bottom. |
Comme Liu and Schneider (2010), cette couche de frottement ne s'exerce pas aux régions équatoriales car à l’extérieur du cylindre tangeant du rayon équatorial, les cylindres convectifs ne coupent pas cette couche et ne subissent pas les frottements liés aux effets de la MHD. | Comme Liu and Schneider (2010), cette couche de frottement ne s'exerce pas aux régions équatoriales car à l’extérieur du cylindre tangeant du rayon équatorial, les cylindres convectifs ne coupent pas cette couche et ne subissent pas les frottements liés aux effets de la MHD. | ||
| Line 174: | Line 158: | ||
Pour le temps (rayleigh_friction_tau), il est similaire à celui utilisé par Liu and Schneider (2010). Elle est fixée à 100 jours terrestres. Mais attention, on fonction de la valeur, la dynamique peut évoluer dans votre simulation. | Pour le temps (rayleigh_friction_tau), il est similaire à celui utilisé par Liu and Schneider (2010). Elle est fixée à 100 jours terrestres. Mais attention, on fonction de la valeur, la dynamique peut évoluer dans votre simulation. | ||
| − | + | == F) Conservation == | |
Dans run_icosa.def: | Dans run_icosa.def: | ||
| − | + | <pre>check_conservation = detailed | |
| − | check_conservation = detailed | + | itau_check_conserv = 320</pre> |
| − | itau_check_conserv = 320 | ||
| − | |||
| − | |||
Le itau_check_conserv est comme itau_physics pour que la contribution de la physique à AAM ne soit pas nulle. de plus il est coûteux d'appeler les diagnostics trop souvent. | Le itau_check_conserv est comme itau_physics pour que la contribution de la physique à AAM ne soit pas nulle. de plus il est coûteux d'appeler les diagnostics trop souvent. | ||
| Line 189: | Line 170: | ||
P. H. Lauritzen, J. T. Bacmeister, T. Dubos, S. Lebonnois, and M. A. Taylor. Held-Suarez simulations with the Community Atmosphere Model Spectral Element (CAM-SE) dynamical core: A global axial angular momentum analysis using Eulerian and floating Lagrangian vertical coordinates. Journal of Advances in Modeling Earth Systems, 6:129-140, 2014. | P. H. Lauritzen, J. T. Bacmeister, T. Dubos, S. Lebonnois, and M. A. Taylor. Held-Suarez simulations with the Community Atmosphere Model Spectral Element (CAM-SE) dynamical core: A global axial angular momentum analysis using Eulerian and floating Lagrangian vertical coordinates. Journal of Advances in Modeling Earth Systems, 6:129-140, 2014. | ||
| − | + | == G) Traceur == | |
| − | + | === - Branche master version du 04/04/2022 dans le dossier Jupiter (traceur) === | |
| − | La version actuelle des fichiers de réglage dans le dossier Jupiter est réglée pour utiliser des traceurs. | + | La version actuelle des fichiers de réglage dans le dossier Jupiter est réglée pour utiliser des traceurs. Si vous souhaiter utiliser le modèle sans traceur, il vous faut modifier les fichiers suivants : |
| − | Si vous souhaiter utiliser le modèle sans traceur, il vous faut modifier les fichiers suivants : | ||
run_icosa.def | run_icosa.def | ||
| − | + | <pre> nqtot = 0</pre> | |
| − | |||
traceur.def | traceur.def | ||
| − | + | <pre> 0</pre> | |
| − | |||
context_dynamico.xml (ligne 125) | context_dynamico.xml (ligne 125) | ||
| − | + | <pre> <field id="q_start" name="q" grid_ref="grid_q_start" prec="8"/> </pre> | |
| + | == H) Bottom du modèle == | ||
| − | + | * refaire tourner le 1D | |
| + | ** changer la pression <code>psurf</code> dans rcm1d.def | ||
| + | ** changer <code>ichoice=1</code> et changer <code>tref</code> (ex: 10b: 330K) | ||
| + | * refaire tourner le 3D (avec makestart) en changeant <code>preff</code> dans <code>[nom_de_la_planète]_const.def</code> | ||
| − | + | = 4. Physical parameterizations = | |
| − | |||
| − | |||
| − | |||
| − | + | == A) Cycle diurne == | |
| − | |||
| − | + | <code>diurnal = .false.</code> Car le temps radiatif est beaucoup plus long sur les planètes géantes. | |
| − | Car le temps radiatif est beaucoup plus long sur les planètes géantes. | ||
| − | + | == B) Anneaux == | |
| − | [Pour saturne uniquement] Il est possible d'ajouter l'ombre des anneaux dans le callphys.def: | + | [Pour saturne uniquement] Il est possible d'ajouter l'ombre des anneaux dans le callphys.def: |
| − | + | <code>rings_shadow = .true.</code> | |
| − | + | == C) Collision-induced absorption data == | |
| − | L'absorption induite par collision est importante dans le transfet radiatif sur les planètes géantes, notamment celle provenant de H2. Cependant, le rapport ortho-para du H2 est différent selon les planètes et peut modifier fortement le chauffage sur ces planètes. | + | L'absorption induite par collision est importante dans le transfet radiatif sur les planètes géantes, notamment celle provenant de H2. Cependant, le rapport ortho-para du H2 est différent selon les planètes et peut modifier fortement le chauffage sur ces planètes. Pour le cas des géantes gazeuses, le rapport ortho-para est normal (rapport 3:1). Mais pour le cas des géantes glacées, le rapport ortho-para est à l'équilibre. Par défaut, le rapport ortho-para est normal (H2orthopara_mixture = normal). Pour les géantes glacées, il faut mettre H2orthopara_mixture = equilibrium . |
| − | Pour le cas des géantes gazeuses, le rapport ortho-para est normal (rapport 3:1). Mais pour le cas des géantes glacées, le rapport ortho-para est à l'équilibre. Par défaut, le rapport ortho-para est normal (H2orthopara_mixture = normal). Pour les géantes glacées, il faut mettre H2orthopara_mixture = equilibrium . | ||
Si vous voulez modifier le fichier utilisé dans un cas de CIA, il faut aller dans le code (dynamico-giant/code/LMDZ.GENERIC/libf/phystd/interpolate????.F90) et changer le nom du fichier (assurez-vous que ce fichier soit dans votre répertoire DATAGENERIC/continuum_data). | Si vous voulez modifier le fichier utilisé dans un cas de CIA, il faut aller dans le code (dynamico-giant/code/LMDZ.GENERIC/libf/phystd/interpolate????.F90) et changer le nom du fichier (assurez-vous que ce fichier soit dans votre répertoire DATAGENERIC/continuum_data). | ||
| Line 236: | Line 212: | ||
Dans la version actuelle, il n'existe pas d'option qui permet de ne pas utiliser certaines contributions de CIA. Par exemple, si votre atmosphère est composé de H2, He et CH4 et que vous ne voulez pas des contributions venant du CH4, il vous faut commenter dans le modèle ces contributions. | Dans la version actuelle, il n'existe pas d'option qui permet de ne pas utiliser certaines contributions de CIA. Par exemple, si votre atmosphère est composé de H2, He et CH4 et que vous ne voulez pas des contributions venant du CH4, il vous faut commenter dans le modèle ces contributions. | ||
| − | + | == D) K-correlated data == | |
Les fichiers k-corrélées doivent se situer dans votre répertoire DATAGENERIC/corrk_data . | Les fichiers k-corrélées doivent se situer dans votre répertoire DATAGENERIC/corrk_data . | ||
| − | + | == E) Cpp mode == | |
| − | cpp_mugaz_mode=0 pour que la valeur de cpp et de mugaz proviennent de la dynamique. | + | cpp_mugaz_mode=0 pour que la valeur de cpp et de mugaz proviennent de la dynamique. cpp_mugaz_mode=1 pour forcer la valeur de cpp et de mugaz dans le callphys.def (à utiliser dans le makestart uniquement) cpp_mugaz_mode=2 pour calculer automatiquement à partir de données de références à 300 K et du gases.def (à éviter) |
| − | cpp_mugaz_mode=1 pour forcer la valeur de cpp et de mugaz dans le callphys.def (à utiliser dans le makestart uniquement) | ||
| − | cpp_mugaz_mode=2 pour calculer automatiquement à partir de données de références à 300 K et du gases.def (à éviter) | ||
| − | + | == F) Generic n-layer aerosols (replaces the former 2-layer and NH3 cloud) == | |
| − | Ce mode permet de créer des couches d'aérosols/nuages ayant une opacité (aeronlay_tauref) à une longueur d'onde donnée (aeronlay_lamref), un rayon de particule fixe (aeronlay_size) situés à une pression fixe et en fonction des propriétés optiques des particules (optprop_aeronlay_vis et optprop_aeronlay_ir). | + | Ce mode permet de créer des couches d'aérosols/nuages ayant une opacité (aeronlay_tauref) à une longueur d'onde donnée (aeronlay_lamref), un rayon de particule fixe (aeronlay_size) situés à une pression fixe et en fonction des propriétés optiques des particules (optprop_aeronlay_vis et optprop_aeronlay_ir). Pour le cas de l'altitude, on peut choisir soit (aeronlay_choice = 1) une pression max (aeronlay_ptop) et une pression min (aeronlay_pbot), soit (aeronlay_choice = 2) une pression min (aeronlay_pbot) et une échelle de hauteur (aeronlay_sclhght). On peut également choisir la variance effective pour les rayons de particules. |
| − | Pour le cas de l'altitude, on peut choisir soit (aeronlay_choice = 1) une pression max (aeronlay_ptop) et une pression min (aeronlay_pbot), soit (aeronlay_choice = 2) une pression min (aeronlay_pbot) et une échelle de hauteur (aeronlay_sclhght). | ||
| − | On peut également choisir la variance effective pour les rayons de particules. | ||
| − | Le nombre de couche de nuage (nlayero) est égal au nombre de scatterers dans la compilation (option -s ). | + | Le nombre de couche de nuage (nlayero) est égal au nombre de scatterers dans la compilation (option -s ). |
Les fichiers de propriétés optiques doivent être dans votre DATAGENERIC. | Les fichiers de propriétés optiques doivent être dans votre DATAGENERIC. | ||
Un exemple pour 3 couches: | Un exemple pour 3 couches: | ||
| − | + | ||
| − | aeronlay = .true. | + | <pre>aeronlay = .true. |
nlayaero = 3 | nlayaero = 3 | ||
aeronlay_tauref = 2.5 0.04 0.1 | aeronlay_tauref = 2.5 0.04 0.1 | ||
| Line 269: | Line 241: | ||
aeronlay_nueff = 0.3 0.3 0.3 | aeronlay_nueff = 0.3 0.3 0.3 | ||
optprop_aeronlay_vis = optprop_aerosol2_vis.dat optprop_aerosol3_vis.dat optprop_carbon4_vis.dat | optprop_aeronlay_vis = optprop_aerosol2_vis.dat optprop_aerosol3_vis.dat optprop_carbon4_vis.dat | ||
| − | optprop_aeronlay_ir = optprop_aerosol2_ir.dat optprop_aerosol3_ir.dat optprop_carbon4_ir.dat | + | optprop_aeronlay_ir = optprop_aerosol2_ir.dat optprop_aerosol3_ir.dat optprop_carbon4_ir.dat</pre> |
| − | + | == G) Panaches thermiques == | |
| − | # | + | # Comme pour toutes nouvelles simulations, il faut commencer par un run 1D de plusieurs décennies permettant d'obtenir un profil de température (<code>temp_profile.txt</code>) et les coefficients ap et bp (<code>apbp.txt</code>) en équilibre radiatif-convectif pour la planète que l'on étudie. Ce run 1D s'effectue dans les dossiers jupiter1d, saturn1d, neptune1d et/ou uranus1d, avec ces options pour le callphys.def : |
| − | + | callrad = true calladj = true tous les autres mots clés à false (y compris <code>calltherm</code>). | |
| − | |||
| − | |||
| − | |||
| − | tous les autres mots clés à false (y compris | ||
| − | 2 | + | <ol start="2" style="list-style-type: decimal;"> |
| − | Il s'agit du run dont l'état initial est le profil de température créé par run 1D ( | + | <li><p>Faire un "makestart" run : permet d'obtenir les fichiers restart à partir du profil de température initial. Il s'agit du run dont l'état initial est le profil de température créé par run 1D (<code>temp_profile.txt</code> est appliquer à chaque point de grille horizontale du modèle). L'état initial est ainsi une planète isotherme horizontalement mais qui varie verticalement. Pour ce run, aucun traceur ne va être utilisé dans le modèle. Néanmoins, il faut renseigner au modèle le nombre de traceurs que nous souhaitons utiliser avec le schéma des panaches thermiques afin qu'il puisse créer la dimension nq et le champ q dans les fichiers <code>restart_icosa.nc</code> et <code>restartfi.nc</code>. dans '''callphys.def''' :</p> |
| − | Pour ce run, aucun traceur ne va être utilisé dans le modèle. Néanmoins, il faut renseigner au modèle le nombre de traceurs que nous souhaitons utiliser avec le schéma des panaches thermiques afin qu'il puisse créer la dimension nq et le champ q dans les fichiers | + | <pre> traceur = true</pre> |
| − | dans | + | <p>dans '''run_icosa.def''' : <code>nqtot = 2</code> (par exemple, h2o_vap et h2o_ice) dans '''traceur.def''' :</p> |
| + | <pre> 2 | ||
| + | h2o_vap | ||
| + | h2o_ice</pre></li> | ||
| + | <li><p>Ajout des quantités pour chaque traceur. Ici, nous ajoutons les profils pour chacun des traceurs dans les fichiers <code>restart_icosa.nc</code> et <code>restartfi.nc</code> "à la main" en utilisant le programme python <code>/processing_codes/tracer_settings.py</code> pour obtenir des fichiers restart avec la bonne abondance d'eau dans le cas présent.</p></li> | ||
| + | <li><p>Commencer la simulation : Il ne reste plus qu'à lancer la simulation 3D avec les nouveaux fichiers restart et les réglages suivant : dans '''callphys.def''' :</p> | ||
| + | <pre> traceur = true | ||
| + | calltherm = true | ||
| + | # thermal plume model options: | ||
| + | divmpl = true | ||
| + | r_aspect_thermals = 2.0 | ||
| + | tau_thermals = 0.0 | ||
| + | betalpha = 0.9 | ||
| + | afact = 0.7 | ||
| + | fact_epsilon = 2.e-4 | ||
| + | alpha_max = 0.7 | ||
| + | fomass_max = 0.5 | ||
| + | pres_limit = 2.e5 | ||
| + | water = true | ||
| + | watercond = true | ||
| + | waterain = true | ||
| + | evap_prec = true</pre></li></ol> | ||
| − | + | dans '''run_icosa.def''' : <code>nqtot = 2</code> dans '''traceur.def''' : | |
| − | dans | ||
| − | dans | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | <pre> 2 | |
| − | + | h2o_vap | |
| + | h2o_ice</pre> | ||
| + | Enfin, ajouter la déclaration et l'écriture des variables relatives à l'utilisation du schéma des thermiques (<code>h2o_vap</code>, <code>h2o_ice</code>, <code>w_plm</code>) dans les fichiers XML de la physique. | ||
| − | + | == H) Rotation == | |
| − | |||
| − | |||
| − | + | Dans [name_of_planet]_const.def, changer le taux de rotation. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | Cela n'a jamais été testé avec 0, mais cela pourrait créer des problèmes (ex: beta). ''vérifier que omega dans saturn_const.def n'intervient pas dans la physique LMDZ.GENERIC/libf/phystd/'' | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | == I) Appel à la physique == | |
| − | + | Il faut changer à ''deux endroits'' en réglant la même valeur: - dans run_icosa.def, changer itau_physics ; - dans run.def, changer iphysiq. Ces paramètres sont exprimés en pas de temps dynamique. Pour appeler la physique à chaque pas de temps dynamique, régler ces paramètres à 1. | |
| − | |||
| − | + | '''Attention: ''' Lorsque l'on change dans le ''run_icosa.def'' la valeur de itau_physics, faire bien attention à la valeur du champ itau_adv (qui controle la frequence d'advection des traceurs par la dynamique, compté en pas de temps dynamique). Il faut que itau_physics soit un multiple de itau_adv. Cela veut dire, que l'on advecte notre champ de traceurs plus souvent (ou autant de fois) que l'on appelle la physique, qui peut transformer ces traceurs. | |
| − | + | = 5. Ecriture des fichiers = | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | == A) Nombre d'écriture dans chaque Xhistins.nc == | |
| − | |||
| − | Prenons l'exemple de l'écriture d'un fichier sur une simu Uranus pour 1000j | + | Prenons l'exemple de l'écriture d'un fichier sur une simu Uranus pour 1000j |
Nombre d'écriture = day_step X ndays/(itau_physics X output_freq) | Nombre d'écriture = day_step X ndays/(itau_physics X output_freq) | ||
| − | + | * ndays = 1000 | |
| − | + | * day_step = 200 | |
| − | + | * itau_physics = 50 | |
| − | + | * output_freq = 200 | |
On a 20 sorties tous les 1000 jours. | On a 20 sorties tous les 1000 jours. | ||
| Line 357: | Line 310: | ||
Globalement, il est conseillé d'avoir 800 à 1000 sorties minimum par année planétaire (voir beaucoup plus en fonction de votre étude). | Globalement, il est conseillé d'avoir 800 à 1000 sorties minimum par année planétaire (voir beaucoup plus en fonction de votre étude). | ||
| − | + | == B) Ajouter une variable == | |
| − | Pour ajouter une variable dans le fichier de sortie Xhistins.nc, il faut l'ajouter dans le field_group correspondant et s'assurer que cette variable est présente en tant que variable de sortie dans dynamico-giant/code/LMDZ.GENERIC/libf/phystd/physiq_mod.F90. Si ce n'est pas le cas, il faut faire un call writediagfi de la variable puis l'ajouter dans un call send_xios_field. | + | Pour ajouter une variable dans le fichier de sortie Xhistins.nc, il faut l'ajouter dans le field_group correspondant et s'assurer que cette variable est présente en tant que variable de sortie dans dynamico-giant/code/LMDZ.GENERIC/libf/phystd/physiq_mod.F90. Si ce n'est pas le cas, il faut faire un call writediagfi de la variable puis l'ajouter dans un call send_xios_field. |
| − | + | == C) Forcer XIOS à écrire tous les .... == | |
| − | Il faut utiliser l'attribut (ici par ex pour forcer l'écriture tous les ts (==time step) ; d'autres délais doivent être possible): | + | Il faut utiliser l'attribut (ici par ex pour forcer l'écriture tous les ts (==time step) ; d'autres délais doivent être possible): <code>sync_freq="1ts"</code> dans le <code><file id=... .... ></code> concerné. |
| − | |||
| − | dans le | ||
| − | + | == D) XIOS server ou client == | |
| − | + | # XIOS client <code>use_server=False</code> Broadwell 24 processeurs sur 28 | |
| − | Broadwell 24 | + | # XIOS server <code>use_server=true</code> Broadwell 24 processurs sur 28 + 4 processeurs pour XIOS |
| − | 2) | + | Le cas 1 est 3 min plus lent que le cas 2 -- sur 2h20...(!) parce qu'on fait peu de sorties. |
| − | |||
| − | + | = 6. Running the model = | |
| − | + | == A) Compilation == | |
| − | |||
Pour compiler le modèle, il suffit d'aller dans le dossier avec vos fichiers .def et .xml et d'utiliser le script compile_occigen.sh si on est sur occigen ou compile_ciclad.sh si on est sur ciclad | Pour compiler le modèle, il suffit d'aller dans le dossier avec vos fichiers .def et .xml et d'utiliser le script compile_occigen.sh si on est sur occigen ou compile_ciclad.sh si on est sur ciclad | ||
| − | + | <code>./compile_occigen.sh</code> | |
| − | Les options qui peuvent être modifiées/ajoutées sont | + | Les options qui peuvent être modifiées/ajoutées sont - -s : le nombre de scatterers - -parallel: mpi ou mpi_omp - -arch: le nom du ficher d'architecture - -arch_path: le chemin vers ce fichier - -job: 8 (conseillé) - -full: compile le code entièrement - -debug: pour trouver un éventuel bug. A ENLEVER obligatoirement s'il n'y a plus de bug car multiplie par 5 votre temp de calcul. |
| − | - -s : le nombre de scatterers | ||
| − | - -parallel: mpi ou mpi_omp | ||
| − | - -arch: le nom du ficher d'architecture | ||
| − | - -arch_path: le chemin vers ce fichier | ||
| − | - -job: 8 (conseillé) | ||
| − | - -full: compile le code entièrement | ||
| − | - -debug: pour trouver un éventuel bug. A ENLEVER obligatoirement s'il n'y a plus de bug car multiplie par 5 votre temp de calcul. | ||
| − | En 1D, d'autres options sont nécessaires: | + | En 1D, d'autres options sont nécessaires: - -t: nombre de traceurs - -d: nombre de niveaux verticaux - -parallel: none (en 1D uniquement) |
| − | - -t: nombre de traceurs | ||
| − | - -d: nombre de niveaux verticaux | ||
| − | - -parallel: none (en 1D uniquement) | ||
| − | + | === - Versions fonctionnelles de dynamico-giant === | |
| − | + | '''Jupiter''' - XIOS revision 1583<br /> | |
| − | - XIOS revision 1583 | + | - ICOSAGCM revision 756<br /> |
| − | - ICOSAGCM revision 756 | + | - IOIPSL revision 339<br /> |
| − | - IOIPSL revision 339 | + | - FCM_V1.2 revision 12<br /> |
| − | - FCM_V1.2 revision 12 | + | - Physics revision 2228 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - Physics revision 2228 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
but check Physics version 2142 and 2180 | but check Physics version 2142 and 2180 | ||
| − | + | '''Saturne (référence Spiga 2020, à vérifier)''' - DYNAMICO --> Revision: 756 - PHYSICS --> Revision: 2005 - XIOS --> Revision: 1583 - IOIPSL --> Revision: 310? | |
| − | - DYNAMICO -- | ||
| − | - PHYSICS -- | ||
| − | - XIOS -- | ||
| − | - IOIPSL -- | ||
| − | + | '''Saturne (simulation de référence sur 61 niveaux, Bardet et al. Icarus 2021)''' - XIOS revision 1583<br /> | |
| − | - XIOS revision 1583 | + | - ICOSAGCM revision 765<br /> |
| − | - ICOSAGCM revision 765 | + | - IOIPSL revision 310<br /> |
| − | - IOIPSL revision 310 | + | - FCM_V1.2 revision 12<br /> |
| − | - FCM_V1.2 revision 12 | + | - Physics revision 2005 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - Physics revision 2005 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | '''Saturne (simulation avec la GWD paramétrisation sur 61 niveaux, chapitre 6 PhD Bardet)''' - XIOS revision 1583<br /> | |
| − | - XIOS revision 1583 | + | - ICOSAGCM revision 765<br /> |
| − | - ICOSAGCM revision 765 | + | - IOIPSL revision 310<br /> |
| − | - IOIPSL revision 310 | + | - FCM_V1.2 revision 12<br /> |
| − | - FCM_V1.2 revision 12 | + | - Physics revision 2213 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - Physics revision 2213 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | '''Saturne (simulation sur 96 niveaux, Bardet et al. Nature Astronomy 2022)''' - XIOS revision 1583<br /> | |
| − | - XIOS revision 1583 | + | - ICOSAGCM revision 765<br /> |
| − | - ICOSAGCM revision 765 | + | - IOIPSL revision 431<br /> |
| − | - IOIPSL revision 431 | + | - FCM_V1.2 revision 12<br /> |
| − | - FCM_V1.2 revision 12 | ||
- Physics revision 2305 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | - Physics revision 2305 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | '''Saturne (simulation avec la GWD paramétrisation sur 96 niveaux, chapitre 6 PhD Bardet)''' - XIOS revision 1583<br /> | |
| − | - XIOS revision 1583 | + | - ICOSAGCM revision 765<br /> |
| − | - ICOSAGCM revision 765 | + | - IOIPSL revision 431<br /> |
| − | - IOIPSL revision 431 | + | - FCM_V1.2 revision 12<br /> |
| − | - FCM_V1.2 revision 12 | + | - Physics revision 2403 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - Physics revision 2403 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | '''Uranus & Neptune (old version)''' - XIOS revision 1944 - ICOSAGCM revision 765 - IOIPSL revision 431 - FCM_V1.2 revision 12<br /> | |
| − | - XIOS revision 1944 | + | - Physics revision 2413 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - ICOSAGCM revision 765 | ||
| − | - IOIPSL revision 431 | ||
| − | - FCM_V1.2 revision 12 | ||
| − | - Physics revision 2413 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | '''Uranus & Neptune (new version)''' - XIOS revision 2203 - ICOSAGCM revision (20/08/2021) - IOIPSL revision 450 - FCM_V1.2 revision 12<br /> | |
| − | - XIOS revision 2203 | + | - Physics revision 2555 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - ICOSAGCM revision (20/08/2021) | ||
| − | - IOIPSL revision 450 | ||
| − | - FCM_V1.2 revision 12 | ||
| − | - Physics revision 2555 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | '''Jupiter, Saturne, Uranus & Neptune [OCCIGEN VERSION]''' - XIOS revision 2319 - ICOSAGCM revision (90f7138a60ebd3644fbbc42bc9dfa22923386385) - IOIPSL revision 453 - FCM_V1.2 revision 12<br /> | |
| − | - XIOS revision 2319 | + | - Physics revision 2655 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - ICOSAGCM revision (90f7138a60ebd3644fbbc42bc9dfa22923386385) | ||
| − | - IOIPSL revision 453 | ||
| − | - FCM_V1.2 revision 12 | ||
| − | - Physics revision 2655 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | '''Jupiter, Saturne, Uranus & Neptune [IRENE VERSION]''' - XIOS revision 2399 - ICOSAGCM revision (4fbd393a9051fd9c1a5b662683f6ad8d0dc2867c) - IOIPSL revision 6234 - FCM_V1.2 revision 12<br /> | |
| − | - XIOS revision 2399 | + | - Physics revision 2842 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) |
| − | - ICOSAGCM revision (4fbd393a9051fd9c1a5b662683f6ad8d0dc2867c) | ||
| − | - IOIPSL revision 6234 | ||
| − | - FCM_V1.2 revision 12 | ||
| − | - Physics revision 2842 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| − | + | == B) Run 1D == | |
Pour une simulation 1D, 1 seul CPU suffit. | Pour une simulation 1D, 1 seul CPU suffit. | ||
| Line 477: | Line 391: | ||
To run on Occigen: | To run on Occigen: | ||
| − | + | <code>sbatch job_mpi</code> | |
To run on Irene: | To run on Irene: | ||
| − | + | <code>ccc_msub job_mpi</code> | |
To see evolution of your job on Occigen: | To see evolution of your job on Occigen: | ||
| − | + | <code>squeue -u username</code> | |
To see evolution of your job on Irene: | To see evolution of your job on Irene: | ||
| − | + | <code>ccc_mpp -u username</code> | |
To kill your job on Occigen: | To kill your job on Occigen: | ||
| − | + | <code>scancel id_of_your_job</code> | |
To kill your job on Irene: | To kill your job on Irene: | ||
| − | + | <code>ccc_mdel id_of_your_job</code> | |
Une fois que votre simulation est fini, lancer le script python printapbp.py qui vous permettra d'obtenir les fichiers apbp.txt et temp_profile.txt nécessaires pour les simulations en 3D. | Une fois que votre simulation est fini, lancer le script python printapbp.py qui vous permettra d'obtenir les fichiers apbp.txt et temp_profile.txt nécessaires pour les simulations en 3D. | ||
| − | + | == C) Run 3D == | |
Pour lancer une première simulation, il faut tout d'abord le faire une seule fois dans le dossier makestart de l'un des dossiers jupiter, saturne, uranus ou neptune. Ce dernier permettra de créer les premiers fichiers restart_icosa.nc et restartfi.nc. Ensuite, il suffit revenir dans le dossier parent (jupiter, saturne, uranus, neptune) et de lancer ses simulations. | Pour lancer une première simulation, il faut tout d'abord le faire une seule fois dans le dossier makestart de l'un des dossiers jupiter, saturne, uranus ou neptune. Ce dernier permettra de créer les premiers fichiers restart_icosa.nc et restartfi.nc. Ensuite, il suffit revenir dans le dossier parent (jupiter, saturne, uranus, neptune) et de lancer ses simulations. | ||
| − | + | === - MPI === | |
| − | Avant de lancer une simulation, il est nécessaire de déterminer | + | Avant de lancer une simulation, il est nécessaire de déterminer - le nombre de procs - le nombre de noeuds |
| − | - le nombre de procs | ||
| − | - le nombre de noeuds | ||
| − | Il faut avant tout savoir qu'il y a 10 x nsplit_i x nsplit_j domaines. | + | Il faut avant tout savoir qu'il y a 10 x nsplit_i x nsplit_j domaines. Ces domaines sont divisés en plusieurs cellules. Il y a (nbp)²/(nsplit_i x nsplit_j) cellules par domaine. Ce nombre est égal au nombre de MPI process. |
| − | Ces domaines sont divisés en plusieurs cellules. Il y a (nbp)²/(nsplit_i x nsplit_j) cellules par domaine. Ce nombre est égal au nombre de MPI process. | ||
| − | Il est important de ne pas faire des tuiles | + | Il est important de ne pas faire des tuiles "trop petites" et donc de garder nsplit_i et nsplit_j tels que nbp/nsplit_{i,j} >=10-15 (plutôt 15). Car il y a des calculs "redondants" faits sur les bords du domaine. Admettons que ton domaine soit 10 x 10 (100 cellules par domaine). Le nombre de cellules au bord du domaine est égal à 2 x nsplit_i + 2 x nsplit_j - 4. Dans ce cas, le nombre de cellules au bord est de 36. Cela signifie que le bord du domaine correspond à 36% du domaine (36 points sur 100). Si le domaine est 15 x 15, alors le bord du domaine n'est plus que de 24.8 %. On aimerait avoir des domaines les plus grands possibles mais il faut aussi voir que chaque cellule correspond à une colonne à résoudre... Et c'est là ou il faut expérimenter un peu pour trouver l'optimum entre le nombre total de proc à employer et le gain effectif (temps total "facturé" au vu du nombre de procs monopolisés pour une simu donnée). Ehouarn |
| − | Ehouarn | ||
Il faut au total que le nombre de procs soit égal à 10 x nsplit_i x nsplit_j qu'il faut répartir entre les noeuds. | Il faut au total que le nombre de procs soit égal à 10 x nsplit_i x nsplit_j qu'il faut répartir entre les noeuds. | ||
| Line 521: | Line 431: | ||
Pour les noeuds, il faut tenir compte qu'un noeud sur Occigen, c'est 24 (ou 28) procs. Disons 24. Quand on demande N procs, le système te donne M noeuds, soit M X 24 procs (les noeuds ne sont pas partagés avec d'autres applications). Et bien sûr on te facturera ces M noeuds, même si tu n'utilises pas tous les procs. Donc il faut s'efforcer de tomber juste et demander un multiple de 24 procs. Même raisonement si tu demandes à utiliser des noeuds de 28 procs. | Pour les noeuds, il faut tenir compte qu'un noeud sur Occigen, c'est 24 (ou 28) procs. Disons 24. Quand on demande N procs, le système te donne M noeuds, soit M X 24 procs (les noeuds ne sont pas partagés avec d'autres applications). Et bien sûr on te facturera ces M noeuds, même si tu n'utilises pas tous les procs. Donc il faut s'efforcer de tomber juste et demander un multiple de 24 procs. Même raisonement si tu demandes à utiliser des noeuds de 28 procs. | ||
| − | Par exemple, nsplit_i = 4 et nsplit_j = 6. 4 x 6 est égal à 24. | + | Par exemple, nsplit_i = 4 et nsplit_j = 6. 4 x 6 est égal à 24. On utilisera donc 10 noeuds pour avoir 240 nprocs. |
| − | On utilisera donc 10 noeuds pour avoir 240 nprocs. | ||
| − | Prenons un autre exemple. nsplit_i = 5 et nsplit_j = 6. 5x6 = 30 | + | Prenons un autre exemple. nsplit_i = 5 et nsplit_j = 6. 5x6 = 30 Or il y a 24 procs par noeuds. Il faudra utiliser 13 noeuds. Et ce ne sont pas 300 procs qui seront facturés au total mais 312. |
| − | Or il y a 24 procs par noeuds. Il faudra utiliser 13 noeuds. Et ce ne sont pas 300 procs qui seront facturés au total mais 312. | ||
Mais comme vu plus haut, en fonction de la configuration de tes domaines, il est possible que le 2nd exemple coûte moins cher en heure CPU que le 1er exemple car il sera plus rapide. | Mais comme vu plus haut, en fonction de la configuration de tes domaines, il est possible que le 2nd exemple coûte moins cher en heure CPU que le 1er exemple car il sera plus rapide. | ||
| − | + | === - OpenMP / MPI === | |
Dans le cas mixte OpenMP/MPI, il est nécessaire de renseigner le nombre de tâche OpenMP et de MPI threads. Dans le script pour le job, on a : | Dans le cas mixte OpenMP/MPI, il est nécessaire de renseigner le nombre de tâche OpenMP et de MPI threads. Dans le script pour le job, on a : | ||
| − | cpus-per-task et OMP_NUM_THREADS correspondent au nombre de tâche OpenMP | + | cpus-per-task et OMP_NUM_THREADS correspondent au nombre de tâche OpenMP ntask-per-node correspond au nombre de tâche par noeud |
| − | ntask-per-node correspond au nombre de tâche par noeud | ||
| − | Pour déterminer la configuration à utiliser en OpenMP/MPI, il faut respecter ces règles: | + | Pour déterminer la configuration à utiliser en OpenMP/MPI, il faut respecter ces règles: - ntask-per-node x nodes correspond au nombre total de MPI threads qu'il devrait y avoir (c'est-à-dire dans le cas MPI seul). - cpus-per-task x ntask-per-node doit être égale ou inférieur au nombre de procs par noeuds (24 ou 28- sur Occigen) - cpus-per-task devrait être ~égal au nombre de niveaux verticaux / 10 - OMP_NUM_THREADS doit être égal au cpus-per-task - omp_level_size (dans le fichier run_icosa.def) devrait être ~égal au nombre de cpus-per-task. (dans le cas MPI seul, il faut le laisser égal à 1) |
| − | - ntask-per-node x nodes correspond au nombre total de MPI threads qu'il devrait y avoir (c'est-à-dire dans le cas MPI seul). | ||
| − | - cpus-per-task x ntask-per-node doit être égale ou inférieur au nombre de procs par noeuds (24 ou 28- sur Occigen) | ||
| − | - cpus-per-task devrait être ~égal au nombre de niveaux verticaux / 10 | ||
| − | - OMP_NUM_THREADS doit être égal au cpus-per-task | ||
| − | - omp_level_size (dans le fichier run_icosa.def) devrait être ~égal au nombre de cpus-per-task. (dans le cas MPI seul, il faut le laisser égal à 1) | ||
Au total, le nombre total de nprocs est égal à cpus-per-task x ntask-per-node x nodes | Au total, le nombre total de nprocs est égal à cpus-per-task x ntask-per-node x nodes | ||
| − | En reprenant le premier exemple MPI (on suppose qu'on a 40 niveaux verticaux), on aura | + | En reprenant le premier exemple MPI (on suppose qu'on a 40 niveaux verticaux), on aura - cpus-per-task = 4 - ntask-per-norde = 6 On a bien cpus-per-task x ntask-per-norde <= 24. Le nombre de noeuds doit être égal à 40 car 6x40 = 240 nprocs MPI Le nombre de procs qu'on utilisera au total sera de 960. |
| − | - cpus-per-task = 4 | ||
| − | - ntask-per-norde = 6 | ||
| − | On a bien cpus-per-task x ntask-per-norde | ||
| − | Le nombre de noeuds doit être égal à 40 car 6x40 = 240 nprocs MPI | ||
| − | Le nombre de procs qu'on utilisera au total sera de 960. | ||
| − | + | === - Comment déterminer le nombre d'heure CPU (consommation en heure)? === | |
Heures CPU = durée_de_la_simu X nombre de procs total facturé | Heures CPU = durée_de_la_simu X nombre de procs total facturé | ||
| Line 558: | Line 455: | ||
Si on veut connaître le nombre d'heure CPU qu'on consommera à partir d'un test, voici le calcul | Si on veut connaître le nombre d'heure CPU qu'on consommera à partir d'un test, voici le calcul | ||
| − | Heures CPU = durée_de_la_simu_test X nombre de procs total facturé X nombre de jour qu'on veut simulé | + | Heures CPU = durée_de_la_simu_test X nombre de procs total facturé X nombre de jour qu'on veut simulé / (nombre de jour simulées pendant la simu test) |
| − | + | == D) Optimisation == | |
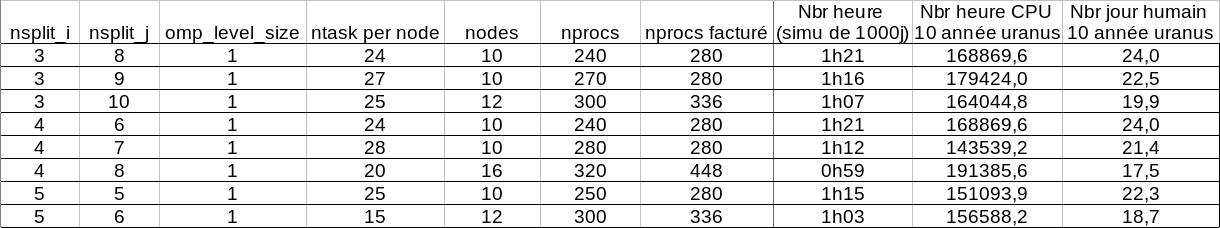
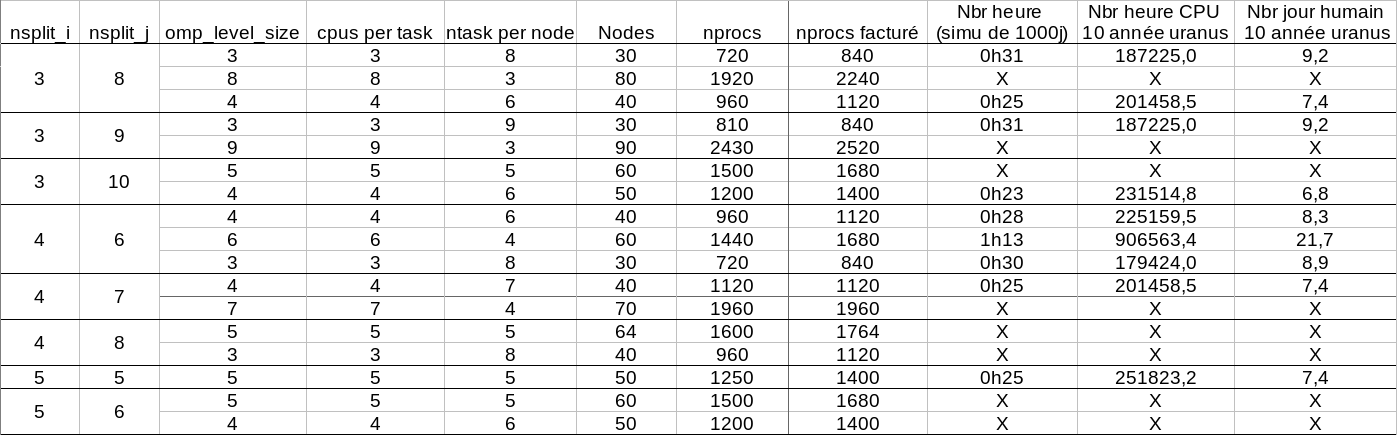
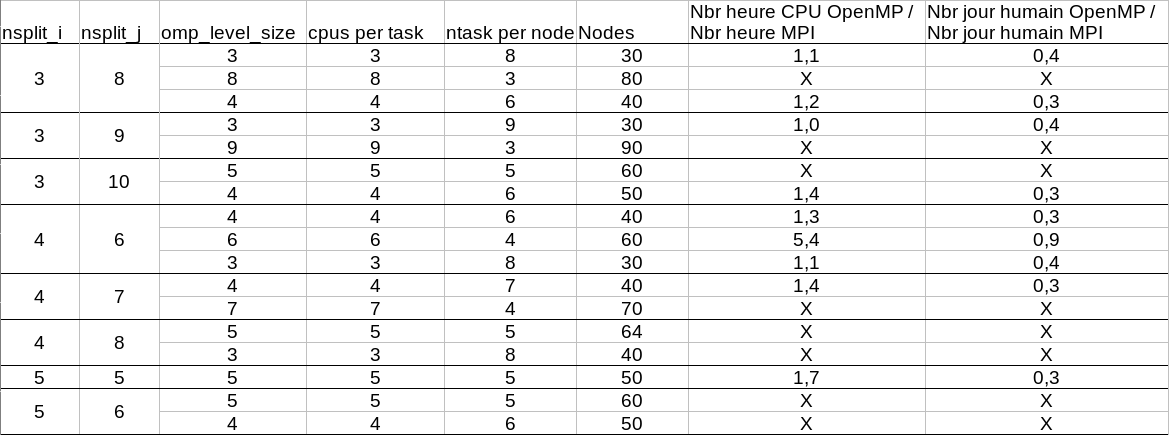
Pour savoir quelle est la meilleure configuration en MPI seul, en OpenMP/MPI et entre les 2, des tests ont été effectués. Ces tests ont été réalisés sur Uranus à nbp80 sur les noeuds Broadwell (28 procs par noeud). 1000 jours uraniens ont été simulés à chaque fois. | Pour savoir quelle est la meilleure configuration en MPI seul, en OpenMP/MPI et entre les 2, des tests ont été effectués. Ces tests ont été réalisés sur Uranus à nbp80 sur les noeuds Broadwell (28 procs par noeud). 1000 jours uraniens ont été simulés à chaque fois. | ||
| − | + | '''MPI:''' [[File:fig1.png]] | |
| − | |||
| − | + | '''OpenMP/MPI:''' [[File:fig2.png]] | |
| − | |||
La comparaison du MPI seul et du mixte OpenMP/MPI permet de tirer la conclusion que le mixte OpenMP/MPI est beaucoup plus rapide (facteur 2.5 à 3) mais consomme plus (facteur 1.1 à 1.7). | La comparaison du MPI seul et du mixte OpenMP/MPI permet de tirer la conclusion que le mixte OpenMP/MPI est beaucoup plus rapide (facteur 2.5 à 3) mais consomme plus (facteur 1.1 à 1.7). | ||
| − | + | [[File:fig3.png]] | |
| − | + | == E) Visualizing the output files == | |
| − | + | === - Comment comparer deux fichiers? file1.nc file2.nc === | |
ncdiff file1.nc file2.nc output.nc | ncdiff file1.nc file2.nc output.nc | ||
| − | + | === - Comment coller plusieurs fichiers file1.nc file2.nc file3.nc === | |
ncrcat file1.nc file2.nc file3.nc output.nc | ncrcat file1.nc file2.nc file3.nc output.nc | ||
| − | Avec juste la vitesse u et v: | + | Avec juste la vitesse u et v: ncrcat -v u -v v file1.nc file2.nc file3.nc output.nc |
| − | ncrcat -v u -v v file1.nc file2.nc file3.nc output.nc | ||
| − | Avec tous les fichiers fileXXX.nc disponibles | + | Avec tous les fichiers fileXXX.nc disponibles ncrcat file*.nc output.nc |
| − | ncrcat file*.nc output.nc | ||
| − | + | === - Comment tracer les champs et divers diagnostics en moyenne zonale? === | |
| − | + | <ul> | |
| − | + | <li>mettre à jour planetoplot et planets</li> | |
| − | + | <li>utiliser precast.py dans planetoplot/examples/ppclass_additional/dynanalysis</li> | |
| − | + | <li><p>changer les options au début (il y a des exemples), exemple pour Saturne</p> | |
| − | fileAP="Xhistins_42.nc" | + | <source lang="python">fileAP="Xhistins_42.nc" |
p_upper,p_lower,nlev = 4.0e2,2.5e5,40 | p_upper,p_lower,nlev = 4.0e2,2.5e5,40 | ||
targetp1d = np.logspace(np.log10(p_lower),np.log10(p_upper),nlev) | targetp1d = np.logspace(np.log10(p_lower),np.log10(p_upper),nlev) | ||
| Line 607: | Line 500: | ||
vartemp = "temperature" | vartemp = "temperature" | ||
outfile = "precast.nc" | outfile = "precast.nc" | ||
| − | nopole = True | + | nopole = True</source></li> |
| − | + | <li>Il faut que apbp.txt soit présent !</li> | |
| − | + | <li><p>Le résultat se trouve dans le fichier indiqué dans outfile.</p></li></ul> | |
| − | |||
| − | + | == F) Debug == | |
| − | + | === - Pour debugger === | |
| − | Réglez | + | Réglez <code>info_level</code> à <code>100</code> dans le fichier <code>iodef.xml</code> . |
| − | + | === - Erreur en début de run, SEGMENTATION FAULT dans la routine <code>advect.f90</code> (ICOSAGCM/ppsrc/transport) au niveau de l'appel à <code>cross_product2</code> === | |
| − | La routine | + | La routine <code>cross_product2</code> effectue des produits vectoriels. Cette erreur vient du fait qu'en essayant de faire des optimisations de calcul, le compilateur se prend "les pieds dans le tapis" et ne sait plus faire le produit vectoriel. Pour empêcher le compilateur de faire des optimisations trop poussées, il faut ajouter l'option <code>-fp-model strict</code> au niveau du mot-clé <code>%BASE_FFLAGS</code> dans tous les fichiers <code>.fcm</code> du modèle (LMDZ.COMMON / ICOSAGCM / ICOSA_LMDZ). |
| − | Pour empêcher le compilateur de faire des optimisations trop poussées, il faut ajouter l'option | ||
| − | + | == G) Pas de radiatif et perturbations == | |
| − | + | <ul> | |
| − | + | <li>faire descendre le modèle</li> | |
| − | + | <li>modifier le code pour perturbations vitesse et pas température</li> | |
| − | + | <li>compiler (voir README.md)</li> | |
| − | + | <li><p>changer dans callphys.def (dans saturn/makestart)</p> | |
| − | surfalbedo = 1.0 | + | <pre>surfalbedo = 1.0 |
| − | surfemis = 0.0 | + | surfemis = 0.0</pre></li> |
| − | + | <li><p>changer dans callphys.def (dans saturn '''et''' saturn/makestart)</p> | |
| − | + | <pre>corrk = .false. | |
| − | |||
| − | corrk = .false. | ||
enertest = .true. | enertest = .true. | ||
| − | randompert = 1 | + | randompert = 1</pre></li> |
| − | `` | + | <li>voir si on garde le flux interne ou non (<code>intheat</code>)</li> |
| − | * voir si on garde le flux interne ou non ( | + | <li>refaire les états initiaux (dans makestart)</li> |
| − | + | <li><p>faire un run court pour voir sur les spectres l'injection d'Ek</p></li></ul> | |
| − | + | ||
| + | == H) PROFILING with DYNAMICO-Giant == | ||
| + | |||
| + | <code>-pg</code>: Generate extra code to write profile information suitable for analysis program <code>gprof</code>. You must use this option when compiling the sources files you want data about and you must '''also use it when linking'''. 1. For DYNAMICO-Giant: in the arch.fcm file of each part of the model (ICOSAGCM, ICOSA_LMDZ, LMDZ.COMMON and XIOS), you have to add the option <code>-pg</code> to <code>%PROD_FFLAGS</code> and <code>%BASE_LD</code> 2. Compile as usual 3. Execute your code as usual 4. Run <code>gprof</code> tool: | ||
| + | |||
| + | <pre> gprof icosa_lmdz.exe goon.out > analysis.txt</pre> | ||
| + | |||
| + | |||
| + | |||
| + | = OLD WIKI (DEPRECATED, JUST FOR REFERENCE/INFORMATION) = | ||
| + | |||
| + | ``Welcome to the dynamico-giant wiki! | ||
| + | |||
| + | == pour debugger == | ||
| + | |||
| + | régler <code>info_level</code> à <code>100</code> dans le fichier <code>iodef.xml</code> | ||
| + | |||
| + | == pourquoi diurnal=.false. == | ||
| + | |||
| + | le temps radiatif est beaucoup plus long sur les géantes | ||
| + | |||
| + | == comment changer la fréquence d'appel à la physique? == | ||
| + | |||
| + | il faut changer à ''deux endroits'' en réglant la même valeur - dans run_icosa.def, changer itau_physics - dans run.def, changer iphysiq ces paramètres sont exprimés en pas de temps dynamique. pour appeler la physique à chaque pas de temps dynamique, régler ces paramètres à 1 | ||
| + | |||
| + | == comment changer la résolution == | ||
| + | |||
| + | [https://github.com/aymeric-spiga/dynamico-giant/commit/f39e3b651c19cefb26da72ab77933520ff8f27f5 voir lien ici] | ||
| + | |||
| + | == comment choisir le nombre de processeurs pour la résolution == | ||
| + | |||
| + | voir commentaires sur run_icosa.def | ||
| + | |||
| + | <pre>########################################### | ||
| + | ## There must be less MPIxOpenMP processes than the 10 x nsplit_i x nsplit_j tiles | ||
| + | ## typically for pure MPI runs, let nproc = 10 x nsplit_i x nsplit_j | ||
| + | ## it is better to have nbp/split >~ 10 | ||
| + | ########################################### | ||
| + | #### 40 noeuds de 24 processeurs = 960 procs | ||
| + | nsplit_i=12 | ||
| + | nsplit_j=8</pre> | ||
| + | Ehouarn: ''Une règle à suivre est de ne pas faire des tuiles "trop petites" et donc de garder nbsplit_i et nbsplit_j tels que nbp/nbsplit >=10-15 (plutôt 15). Car il y a des calculs "redondants" faits sur les bords du domaine. Or si ton domaine est 10''10, le bord du domaine c'est 38% du domaine (38 points sur le bord pour 100 points en tout) alors que si ton domaine est 15''15, le bord du domaine n'est plus que 56/(15''15) = 25.7%. 56=2''15+2''13 (pour ne pas compter 2 fois les coins). On voudrait du coup des domaine les plus grand possible, clairement, mais il faut aussi voir que chaque domaine c'est nbp''nbp colonnes à résoudre sur ce même proc... Et c'est là ou il faut expérimenter un peu pour trouver l'optimum entre le nombre total de proc à employer et le gain effectif (temps total "facturé" au vu du nombre de procs monopolisés pour une simu donnée).'' | ||
| + | |||
| + | _Pour les noeuds, il faut tenir compte qu'un noeud sur Occigen, c'est 24 (ou 28) procs. Disons 24. Quand on demande N procs, le système te donne M noeuds, soit M*24 procs (les noeuds ne sont pas partagés avec d'autres applications). Et bien sûr on te facturera ces M noeuds, même si tu n'utilises pas tous les procs. Donc il faut s'efforcer de tomber juste et demander un multiple de 24 procs. Même raisonement si tu demandes à utiliser des noeuds de 28 procs._ | ||
| + | |||
| + | == comment changer la rotation? == | ||
| + | |||
| + | dans saturn_const.def, changer le taux de rotation jamais testé avec 0, mais pourrait créer des problèmes (ex: beta) ''vérifier que omega dans saturn_const.def n'intervient pas dans la physique LMDZ.GENERIC/libf/phystd/'' | ||
| + | |||
| + | == la dissipation c'est où? == | ||
| + | |||
| + | laplacien itéré: hyperviscosity - tau_graddiv,tau_gradrot,tau_divgrad: periode (plus c'est petit, plus la dissipation est efficace) - nitergdiv,nitergrot,niterdivgrad: ordre du laplacien (plus c'est grand, plus on dissipe sélectivement les petites échelles) | ||
| + | |||
| + | == comment changer la grille cible du remapping on-the-fly == | ||
| + | |||
| + | dans context_lmdz_physics.xml changer les paramètres ni_glo et nj_glo par exemple pour remapper sur du lat/lon à 360 pts en latitude et 720 pts en longitude (0.5°) <domain id="dom_regular" ni_glo="720" nj_glo="360" type="rectilinear"> | ||
| + | |||
| + | == Forcer XIOS à écrire tous les .... == | ||
| + | |||
| + | Il faut utiliser l'attribut (ici par ex pour forcer l'écriture tous les ts (==time step) ; d'autres délais doivent être possible): <code>sync_freq="1ts"</code> dans le <code><file id=... .... ></code> concerné. | ||
| + | |||
| + | == comment changer le bottom du modele == | ||
| + | |||
| + | * refaire tourner le 1D | ||
| + | ** changer la pression <code>psurf</code> dans rcm1d.def | ||
| + | ** changer <code>ichoice=1</code> et changer <code>tref</code> (ex: 10b: 330K) | ||
| + | * refaire tourner le 3D (avec makestart) en changeant <code>preff</code> dans <code>saturn_const.def</code> | ||
| + | |||
| + | == comment changer la fréquence de sortie des Xhistins.nc == | ||
| + | |||
| + | les commandes XIOS sont appelées depuis la physique (dans la config présente) dans context_lmdz_physics.xml il suffit de changer output_freq ''attention'' ts se comprend comme le pas de temps physique (voir donc dans run_icosa.def les paramètres dt et itau_physics pour le connaître) par exemple, pour des sorties tous les 20 jours Saturne | ||
| + | |||
| + | <source lang="xml"><file id="histins" | ||
| + | name="Xhistins" | ||
| + | output_freq="40ts" | ||
| + | type="one_file" | ||
| + | enabled=".true."></source> | ||
| + | == pas de radiatif et perturbations == | ||
| + | |||
| + | <ul> | ||
| + | <li>faire descendre le modèle</li> | ||
| + | <li>modifier le code pour perturbations vitesse et pas température</li> | ||
| + | <li>compiler (voir README.md)</li> | ||
| + | <li><p>changer dans callphys.def (dans saturn/makestart)</p> | ||
| + | <pre>surfalbedo = 1.0 | ||
| + | surfemis = 0.0</pre></li> | ||
| + | <li><p>changer dans callphys.def (dans saturn '''et''' saturn/makestart)</p> | ||
| + | <pre>corrk = .false. | ||
| + | enertest = .true. | ||
| + | randompert = 1</pre></li> | ||
| + | <li>voir si on garde le flux interne ou non (<code>intheat</code>)</li> | ||
| + | <li>refaire les états initiaux (dans makestart)</li> | ||
| + | <li><p>faire un run court pour voir sur les spectres l'injection d'Ek</p></li></ul> | ||
| + | |||
| + | == comment comparer deux fichiers? file1.nc file2.nc == | ||
| + | |||
| + | ncdiff file1.nc file2.nc output.nc | ||
| + | |||
| + | == comment coller plusieurs fichiers file1.nc file2.nc file3.nc == | ||
| + | |||
| + | ncrcat file1.nc file2.nc file3.nc output.nc avec juste la vitesse u et v ncrcat -v u -v v file1.nc file2.nc file3.nc output.nc avec tous les fichiers fileXXX.nc disponibles ncrcat file*.nc output.nc | ||
| + | |||
| + | == comment tracer les champs et divers diagnostics en moyenne zonale? == | ||
| + | |||
| + | <ul> | ||
| + | <li>mettre à jour planetoplot et planets</li> | ||
| + | <li>utiliser precast.py dans planetoplot/examples/ppclass_additional/dynanalysis</li> | ||
| + | <li><p>changer les options au début (il y a des exemples), exemple pour Saturne</p> | ||
| + | <source lang="python">fileAP="Xhistins_42.nc" | ||
| + | p_upper,p_lower,nlev = 4.0e2,2.5e5,40 | ||
| + | targetp1d = np.logspace(np.log10(p_lower),np.log10(p_upper),nlev) | ||
| + | myp = planets.Saturn | ||
| + | day_per_year = 24430. | ||
| + | short = False | ||
| + | includels = False | ||
| + | charx = "0,360" | ||
| + | ispressure = False | ||
| + | vartemp = "temperature" | ||
| + | outfile = "precast.nc" | ||
| + | nopole = True</source></li> | ||
| + | <li>Il faut que apbp.txt soit présent !</li> | ||
| + | <li><p>le résultat se trouve dans le fichier indiqué dans outfile</p></li></ul> | ||
| + | |||
| + | == bilan de moment cinétique == | ||
| + | |||
| + | dans run_icosa.def | ||
| + | |||
| + | <pre>check_conservation = detailed | ||
| + | itau_check_conserv = 160</pre> | ||
| + | le itau_check_conserv est comme itau_physics pour que la contribution de la physique à AAM ne soit pas nulle. de plus il est coûteux d'appeler les diagnostics trop souvent. | ||
| + | |||
| + | S. Lebonnois, C. Covey, A. Grossman, H. Parish, G. Schubert, R. Walterscheid, P. Lauritzen, and C. Jablonowski. Angular momentum budget in General Circulation Models of superrotating atmospheres: A critical diagnostic. Journal of Geophysical Research (Planets), 117:E12004, 2012. | ||
| + | |||
| + | P. H. Lauritzen, J. T. Bacmeister, T. Dubos, S. Lebonnois, and M. A. Taylor. Held-Suarez simulations with the Community Atmosphere Model Spectral Element (CAM-SE) dynamical core: A global axial angular momentum analysis using Eulerian and floating Lagrangian vertical coordinates. Journal of Advances in Modeling Earth Systems, 6:129-140, 2014. | ||
| + | |||
| + | == XIOS server ou client == | ||
| + | |||
| + | # XIOS client <code>use_server=False</code> Broadwell 24 processeurs sur 28 | ||
| + | # XIOS server <code>use_server=true</code> Broadwell 24 processurs sur 28 + 4 processeurs pour XIOS | ||
| + | |||
| + | cas 1 est 3 min plus lent que cas 2 -- sur 2h20...! parce qu'on fait peu de sorties | ||
| + | |||
| + | == Optimisation == | ||
| + | |||
| + | # 40 noeuds au lieu de 50 noeuds, même temps de calcul...! Testé dans la branche https://github.com/aymeric-spiga/dynamico-giant/tree/work_61levels | ||
| + | # <code>sync_freq=40ts</code> (ou multiple de, si 40ts est la fréquence d'écriture) dans context_lmdz_physics.xml doit être inclus sinon il n'est pas réglé et XIOS transfère tout à la fin du run ce qui prend du temps (et peut occasionner des problèmes de mémoire) | ||
| + | |||
| + | == Versions fonctionnelles de dynamico-giant == | ||
| + | |||
| + | === Jupiter === | ||
| + | |||
| + | * XIOS revision 1583<br /> | ||
| + | |||
| + | * ICOSAGCM revision 756<br /> | ||
| + | |||
| + | * IOIPSL revision 339<br /> | ||
| + | |||
| + | * FCM_V1.2 revision 12<br /> | ||
| + | |||
| + | * Physics revision 2228 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| + | |||
| + | but check Physics version 2142 and 2180 | ||
| + | |||
| + | === Saturne (référence Spiga 2020, à vérifier) === | ||
| + | |||
| + | * DYNAMICO --> Revision: 756 | ||
| + | * PHYSICS --> Revision: 2005 | ||
| + | * XIOS --> Revision: 1583 | ||
| + | * IOIPSL --> Revision: 310? | ||
| + | |||
| + | === Saturne (simulation de référence sur 61 niveaux, Bardet et al. Icarus 2021) === | ||
| + | |||
| + | * XIOS revision 1583<br /> | ||
| + | |||
| + | * ICOSAGCM revision 765<br /> | ||
| + | |||
| + | * IOIPSL revision 310<br /> | ||
| + | |||
| + | * FCM_V1.2 revision 12<br /> | ||
| + | |||
| + | * Physics revision 2005 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| + | |||
| + | === Saturne (simulation avec la GWD paramétrisation sur 61 niveaux, chapitre 6 PhD Bardet) === | ||
| + | |||
| + | * XIOS revision 1583<br /> | ||
| + | |||
| + | * ICOSAGCM revision 765<br /> | ||
| + | |||
| + | * IOIPSL revision 310<br /> | ||
| + | |||
| + | * FCM_V1.2 revision 12<br /> | ||
| + | |||
| + | * Physics revision 2213 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| + | |||
| + | === Saturne (simulation sur 96 niveaux, Bardet et al. Nature Astronomy 2022) === | ||
| + | |||
| + | * XIOS revision 1583<br /> | ||
| + | |||
| + | * ICOSAGCM revision 765<br /> | ||
| + | |||
| + | * IOIPSL revision 431<br /> | ||
| + | |||
| + | * FCM_V1.2 revision 12<br /> | ||
| + | |||
| + | * Physics revision 2305 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| + | |||
| + | === Saturne (simulation avec la GWD paramétrisation sur 96 niveaux, chapitre 6 PhD Bardet) === | ||
| + | |||
| + | * XIOS revision 1583<br /> | ||
| + | |||
| + | * ICOSAGCM revision 765<br /> | ||
| + | |||
| + | * IOIPSL revision 431<br /> | ||
| + | |||
| + | * FCM_V1.2 revision 12<br /> | ||
| + | |||
| + | * Physics revision 2403 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| + | |||
| + | === Uranus & Neptune (old version) === | ||
| + | |||
| + | * XIOS revision 1944 | ||
| + | * ICOSAGCM revision 765 | ||
| + | * IOIPSL revision 431 | ||
| + | * FCM_V1.2 revision 12<br /> | ||
| + | |||
| + | * Physics revision 2413 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| + | |||
| + | === Uranus & Neptune (new version) === | ||
| + | |||
| + | * XIOS revision 2203 | ||
| + | * ICOSAGCM revision (20/08/2021) | ||
| + | * IOIPSL revision 450 | ||
| + | * FCM_V1.2 revision 12<br /> | ||
| + | |||
| + | * Physics revision 2555 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH) | ||
| + | |||
| + | works on 03/03/2022 for all HEAD | ||
| + | |||
| + | == Les fichiers d'architecture (Installation sur un nouveau cluster) == | ||
| + | |||
| + | En pratique LMDZ.COMMON, ICOSA_LMDZ et IOIPSL peuvent utiliser exactement le même fichier arch.fcm ; mais celui pour ICOSAGCM est légèrement différent (les %FPP_DEF diffèrent, peut-être aussi le %FPP). | ||
| + | |||
| + | make_icosa_lmdz doit être lancé avec -arch_path ../ARCH puisque les arch.env et arch.path communs se trouvent dans ../ARCH (l'option -arch_path ne concerne d'ailleurs que les fichiers arch.env et arch.path; le fichier arch.fcm recherché sera toujours celui dans le "arch" de chacun des modèles. Donc il faut bien mettre pour chacun des quatre modèles (LMDZ.COMMON, ICOSA_LMDZ,IOIPSL et ICOSAGCM) le arch.fcm dans le sous-dossier arch/ du modèle correspondant. | ||
| + | |||
| + | XIOS est écrit en C++, et pas en Fortran. Le fichier arch.fcm correspondant est donc nécessairement différent de celui des quatre autres modèles. Pour créér ce arch.fcm, prendre exemple sur les fichiers .fcm déjà présent dans XIOS/arch/, avec une architecture similaire à celle du nouveau cluster. En particulier, il faut utiliser des versions de gcc/gfortran > 6+. Il faut absolument avoir une bibliothèque HDF5 compilée en parallèle, ainsi que netcdf-C et netcdf-fortran (et peut être aussi netcdf-Cxx) compilé en parallèle. Sans ça, il sera peut-être possible de compiler le modèle, mais pas de lancer une simulation en utilisant XIOS. | ||
| + | |||
| + | == Commencer une nouvelle simulation en utilisant le schéma des panaches thermiques == | ||
| + | |||
| + | # Comme pour toutes nouvelles simulations, il faut commencer par un run 1D de plusieurs décennies permettant d'obtenir un profil de température (<code>temp_profile.txt</code>) et les coefficients ap et bp (<code>apbp.txt</code>) en équilibre radiatif-convectif pour la planète que l'on étudie. Ce run 1D s'effectue dans les dossiers jupiter1d, saturn1d, neptune1d et/ou uranus1d, avec ces options pour le callphys.def : | ||
| + | |||
| + | callrad = true calladj = true tous les autres mots clés à false (y compris <code>calltherm</code>). | ||
| + | |||
| + | <ol start="2" style="list-style-type: decimal;"> | ||
| + | <li><p>Faire un "makestart" run : permet d'obtenir les fichiers restart à partir du profil de température initial. Il s'agit du run dont l'état initial est le profil de température créé par run 1D (<code>temp_profile.txt</code> est appliquer à chaque point de grille horizontale du modèle). L'état initial est ainsi une planète isotherme horizontalement mais qui varie verticalement. Pour ce run, aucun traceur ne va être utilisé dans le modèle. Néanmoins, il faut renseigner au modèle le nombre de traceurs que nous souhaitons utiliser avec le schéma des panaches thermiques afin qu'il puisse créer la dimension nq et le champ q dans les fichiers <code>restart_icosa.nc</code> et <code>restartfi.nc</code>. dans '''callphys.def''' :</p> | ||
| + | <pre> traceur = true</pre> | ||
| + | <p>dans '''run_icosa.def''' : <code>nqtot = 2</code> (par exemple, h2o_vap et h2o_ice) dans '''traceur.def''' :</p> | ||
| + | <pre> 2 | ||
| + | h2o_vap | ||
| + | h2o_ice</pre></li> | ||
| + | <li><p>Ajout des quantités pour chaque traceur. Ici, nous ajoutons les profils pour chacun des traceurs dans les fichiers <code>restart_icosa.nc</code> et <code>restartfi.nc</code> "à la main" en utilisant le programme python <code>/processing_codes/tracer_settings.py</code> pour obtenir des fichiers restart avec la bonne abondance d'eau dans le cas présent.</p></li> | ||
| + | <li><p>Commencer la simulation : Il ne reste plus qu'à lancer la simulation 3D avec les nouveaux fichiers restart et les réglages suivant : dans '''callphys.def''' :</p> | ||
| + | <pre> traceur = true | ||
| + | calltherm = true | ||
| + | # thermal plume model options: | ||
| + | divmpl = true | ||
| + | r_aspect_thermals = 2.0 | ||
| + | tau_thermals = 0.0 | ||
| + | betalpha = 0.9 | ||
| + | afact = 0.7 | ||
| + | fact_epsilon = 2.e-4 | ||
| + | alpha_max = 0.7 | ||
| + | fomass_max = 0.5 | ||
| + | pres_limit = 2.e5 | ||
| + | water = true | ||
| + | watercond = true | ||
| + | waterain = true | ||
| + | evap_prec = true</pre></li></ol> | ||
| + | |||
| + | dans '''run_icosa.def''' : <code>nqtot = 2</code> dans '''traceur.def''' : | ||
| + | |||
| + | <pre> 2 | ||
| + | h2o_vap | ||
| + | h2o_ice</pre> | ||
| + | Enfin, ajouter la déclaration et l'écriture des variables relatives à l'utilisation du schéma des thermiques (<code>h2o_vap</code>, <code>h2o_ice</code>, <code>w_plm</code>) dans les fichiers XML de la physique. | ||
| + | |||
| + | == Erreur en début de run, SEGMENTATION FAULT dans la routine <code>advect.f90</code> (ICOSAGCM/ppsrc/transport) au niveau de l'appel à <code>cross_product2</code>. == | ||
| + | |||
| + | La routine <code>cross_product2</code> effectue des produits vectoriels. Cette erreur vient du fait qu'en essayant de faire des optimisations de calcul, le compilateur se prend "les pieds dans le tapis" et ne sait plus faire le produit vectoriel. Pour empêcher le compilateur de faire des optimisations trop poussées, il faut ajouter l'option <code>-fp-model strict</code> au niveau du mot-clé <code>%BASE_FFLAGS</code> dans tous les fichiers <code>.fcm</code> du modèle (LMDZ.COMMON / ICOSAGCM / ICOSA_LMDZ) | ||
| + | |||
| + | == Branche master version du 04/04/2022 dans le dossier Jupiter == | ||
| + | |||
| + | La version actuelle des fichiers de réglage dans le dossier Jupiter sont réglés pour utiliser des traceurs. Si vous souhaiter utiliser le modèle sans traceur, il vous faut modifier les fichiers suivants : | ||
| + | |||
| + | run_icosa.def | ||
| + | |||
| + | <pre> nqtot = 0</pre> | ||
| + | traceur.def | ||
| + | |||
| + | <pre> 0</pre> | ||
| + | context_dynamico.xml (ligne 125) | ||
| + | |||
| + | <pre> <field id="q_start" name="q" grid_ref="grid_q_start" prec="8"/> </pre> | ||
| + | == PROFILING with DYNAMICO-Giant: == | ||
| + | |||
| + | <code>-pg</code>: Generate extra code to write profile information suitable for analysis program <code>gprof</code>. You must use this option when compiling the sources files you want data about and you must '''also use it when linking'''. 1. For DYNAMICO-Giant: in the arch.fcm file of each part of the model (ICOSAGCM, ICOSA_LMDZ, LMDZ.COMMON and XIOS), you have to add the option <code>-pg</code> to <code>%PROD_FFLAGS</code> and <code>%BASE_LD</code> 2. Compile as usual 3. Execute your code as usual 4. Run <code>gprof</code> tool: | ||
| − | + | <pre> gprof icosa_lmdz.exe goon.out > analysis.txt</pre> | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | [[Category:DYNAMICO]] | |
| + | [[Category:Generic-DYNAMICO]] | ||
Latest revision as of 14:05, 18 October 2024
[transferred from github, now active here]
Contents
- 1 1. Introduction
- 2 2. Installation
- 3 3. General parameters
- 4 4. Physical parameterizations
- 5 5. Ecriture des fichiers
- 6 6. Running the model
- 7 OLD WIKI (DEPRECATED, JUST FOR REFERENCE/INFORMATION)
- 7.1 pour debugger
- 7.2 pourquoi diurnal=.false.
- 7.3 comment changer la fréquence d'appel à la physique?
- 7.4 comment changer la résolution
- 7.5 comment choisir le nombre de processeurs pour la résolution
- 7.6 comment changer la rotation?
- 7.7 la dissipation c'est où?
- 7.8 comment changer la grille cible du remapping on-the-fly
- 7.9 Forcer XIOS à écrire tous les ....
- 7.10 comment changer le bottom du modele
- 7.11 comment changer la fréquence de sortie des Xhistins.nc
- 7.12 pas de radiatif et perturbations
- 7.13 comment comparer deux fichiers? file1.nc file2.nc
- 7.14 comment coller plusieurs fichiers file1.nc file2.nc file3.nc
- 7.15 comment tracer les champs et divers diagnostics en moyenne zonale?
- 7.16 bilan de moment cinétique
- 7.17 XIOS server ou client
- 7.18 Optimisation
- 7.19 Versions fonctionnelles de dynamico-giant
- 7.19.1 Jupiter
- 7.19.2 Saturne (référence Spiga 2020, à vérifier)
- 7.19.3 Saturne (simulation de référence sur 61 niveaux, Bardet et al. Icarus 2021)
- 7.19.4 Saturne (simulation avec la GWD paramétrisation sur 61 niveaux, chapitre 6 PhD Bardet)
- 7.19.5 Saturne (simulation sur 96 niveaux, Bardet et al. Nature Astronomy 2022)
- 7.19.6 Saturne (simulation avec la GWD paramétrisation sur 96 niveaux, chapitre 6 PhD Bardet)
- 7.19.7 Uranus & Neptune (old version)
- 7.19.8 Uranus & Neptune (new version)
- 7.20 Les fichiers d'architecture (Installation sur un nouveau cluster)
- 7.21 Commencer une nouvelle simulation en utilisant le schéma des panaches thermiques
- 7.22 Erreur en début de run, SEGMENTATION FAULT dans la routine advect.f90 (ICOSAGCM/ppsrc/transport) au niveau de l'appel à cross_product2.
- 7.23 Branche master version du 04/04/2022 dans le dossier Jupiter
- 7.24 PROFILING with DYNAMICO-Giant:
1. Introduction
"Welcome to the dynamico-giant wiki"
Dynamico-giant est un dépôt git qui permet d'installer le modèle Dynamico avec les fichiers de configs propres aux planètes géantes gazeuses et géantes glacées. Ci-dessous, vous trouverez presque (car nous ne sommes pas parfaits) toutes les informations sur l'installation, la compilation du modèle et les paramètres que vous pouvez modifier pour faire tourner le modèle.
"Allons-y!"
2. Installation
A) Fichiers d'architecture
- Modules
Before installation, set environment (do it once) in your .bash_profile.
Modules used on Occigen:
ulimit -s unlimited # modules module purge module load intel/17.0 module load intelmpi/2017.0.098 module load hdf5/1.8.17 module load netcdf/4.4.0_fortran-4.4.2 module load ncview module load nco module load qt module load python
Modules used on Ciclad:
ulimit -s unlimited # modules module purge module load gnu/4.9.3 module load intel/15.0.6.233 module load openmpi/1.6.5-ifort module load hdf5/1.8.18-parallel-ifort module load netcdf4/4.4.1.1-parallel-ifort
Beware, if you change version of model (newer or older version), it's possible that you have to change modules...
- Installation sur un nouveau cluster
En pratique LMDZ.COMMON, ICOSA_LMDZ et IOIPSL peuvent utiliser exactement le même fichier arch.fcm ; mais celui pour ICOSAGCM est légèrement différent (les %FPP_DEF diffèrent, peut-être aussi le %FPP).
make_icosa_lmdz doit être lancé avec -arch_path ../ARCH puisque les arch.env et arch.path communs se trouvent dans ../ARCH (l'option -arch_path ne concerne d'ailleurs que les fichiers arch.env et arch.path; le fichier arch.fcm recherché sera toujours celui dans le "arch" de chacun des modèles. Donc il faut bien mettre pour chacun des quatre modèles (LMDZ.COMMON, ICOSA_LMDZ,IOIPSL et ICOSAGCM) le arch.fcm dans le sous-dossier arch/ du modèle correspondant.
L'installation d'IOIPSL doit se faire à partir du bon script bash. Si vous utilisez un server autre qu'occigen, il vous faut changer le nom du script à utiliser dans install_ioipsl.sh. Par exemple, il vous faudra utiliser install_ioipsl_ciclad-ifort.bash pour CICLAD.
XIOS est écrit en C++, et pas en Fortran. Le fichier arch.fcm correspondant est donc nécessairement différent de celui des quatre autres modèles. Pour créér ce arch.fcm, prendre exemple sur les fichiers .fcm déjà présent dans XIOS/arch/, avec une architecture similaire à celle du nouveau cluster. En particulier, il faut utiliser des versions de gcc/gfortran > 6+. Il faut absolument avoir une bibliothèque HDF5 compilée en parallèle, ainsi que netcdf-C et netcdf-fortran (et peut être aussi netcdf-Cxx) compilé en parallèle. Sans ça, il sera peut-être possible de compiler le modèle, mais pas de lancer une simulation en utilisant XIOS.
B. Download
Download structure
Take place on a repository and git clone the model.
On occigen:
cd $SCRATCHDIR git clone https://github.com/aymeric-spiga/dynamico-giant.git [optional different name]
C. Install
Install code
cd dynamico-giant ./install.sh ./install_ioipsl.sh
A login and a password are necessary to install IOIPSL (only for the first time, after that we can save them). Please contact TGCC computing center to get the login/password.
After that, please add "$PWD"/FCM_V1.2/bin/ to PATH environment variable.
Ant that's all, we can now change parameters of files or compile the code to do a test (part 6) or eat a beautiful tartiflette (can be also do during compilation).
3. General parameters
A) Mesh grid & resolution
On run_icosa.def:
nbp --> number of subdivision on a main triangle: integer (default=40)
nbp = sqrt((nbr_lat x nbr_lon)/10)
nbp 20 40 80 160
T-edge length (km) 500 250 120 60
Example: nbp(128x96)=35 -> 40
nbp(256x192)=70 -> 80
nbp(360x720)=160 -> 160
nsplit_i, nsplit_j --> sub splitting of main rhombus: integer
Example: for nbp=80, nsplit_i=4,nsplit_j=6
nbp/nsplit_{i,j} = 20 > 10 & 13 > 10 --> GOOD
- Remapping
Pour le remapping, il faut aller dans context_lmdz_physics.xml changer les paramètres ni_glo et nj_glo par exemple pour remapper sur du lat/lon à 360 pts en latitude et 720 pts en longitude (0.5°).
<domain id="dom_regular" ni_glo="720" nj_glo="360" type="rectilinear">
B) Time
- ndays is number of days to simulate.
- day_step is number of dynamical time step per day
- Dynamics called every day_length(in s) / day_step per day
- 1 ts (physical timestep) = (day_length(in s)/day_step) x itau_physics
- Physics called (day_step / itau_physics) per day
- Physics called day_length(in s) / ts per day
- Radiative called every Physics x iradia physical timestep
- Radiative called every iradia/Physics days
C) Sponge layer
iflag_sponge=0 for no sponge (default)
iflag_sponge=1 for sponge over 4 topmost layers
iflag_sponge=2 for sponge from top to ~1% of top layer pressure
mode_sponge=1 for u,v --> 0
mode_sponge=2 for u,v --> zonal mean
mode_sponge=3 for u,v,h --> zonal mean
tau_sponge --> damping frequency at last layer
--> e-5 medium / e-4 strong yet reasonable / e-3 very strong
Spiga et al (2020): Shaw and Shepherd (2007) showed that the inclusion of sponge-layer parameterizations that do not conserve angular momentum (which is the case for Rayleigh drag), or allow for momentum to escape to space, implies a sensitivity of the dynamical results (especially zonal wind speed) to the choice for model top or drag characteristic timescale, because of spurious downward influence when momentum conservation is violated.
Il ne faut donc pas ajouter de sponge layer pour les planètes géantes (iflag_sponge = 0)
D) Dissipation
Spiga et al (2020): A subgrid-scale dissipation term is included in our Saturn DYNAMICO GCM to prevent the accumulation of energy at scales close to the grid resolution, caused by the GCM not resolving the turbulent scales at which this energy is dissipated. This hyperviscosity term is written in our Saturn DYNAMICO model as an iterated Laplacian term on a given variable. The three variables denoted are vorticity, divergence, and potential temperature, chosen to set horizontal dissipation on respectively the rotational component of the flow (e.g. Rossby waves), the divergent component of the flow (e.g. gravity waves), and the diabatic perturbations (e.g. coming from the physical packages).
tau_graddiv --> dissipation timescale of smallest wvl: u,v (gradiv) : real (default=5000) tau_gradrot --> dissipation timescale of smallest wvl: u,v (nxgradrot) : real (default=5000) tau_divgrad --> dissipation timescale of smallest wvl: h (divgrad) : real (default=5000) nitergdiv --> number of iterations for gradiv operator : integer (default=1) nitergrot --> number of iterations for nxgradrot operator : integer (default=1) niterdivgrad --> number of iterations for divgrad operator : integer (default=1)
tau_graddiv,tau_gradrot,tau_divgrad correspondent au temps de dissipation (plus c'est petit, plus la dissipation est efficace). nitergdiv,nitergrot,niterdivgrad correspondent à l'ordre du laplacien (plus c'est grand, plus on dissipe sélectivement les petites échelles).
Attention, Trop dissiper -> instabilité numérique
Pas assez dissiper -> instabilité dynamique trop forte
L'ordre du laplacien dans les fichiers .def est suffisant. S'il faut changer la dissipation, il vaut mieux changer les temps de dissipation que l'ordre du laplacien (risque d'instabilité numérique).
- Facteurs de dissipation
A partir de ces valeurs de temps de dissipation, il est également possible d'ajouter 2 facteurs de dissipation (fac_mid et fac_up) qui permettent d'augmenter la dissipation à partir de certains niveaux. Le fac_mid permet de diminuer le temps de dissipation d'un facteur sur toute l'atmosphère jusqu'au bottom. Il y a une zone transition de quelques niveaux entre le bottom (le temp de dissipation au bottom est sans facteur) et quelques niveaux au-dessus où la valeur du temps de dissipation est avec ce facteur jusqu'au top. On ne peut donc pas choisir l'altitude et la zone de transition avec ce facteur. Quant au fac_up, il permet de diminuer le temps de dissipation d'un facteur à partir d'une altitude et d'une épaisseur de zone de transition qu'on peut choisir. On peut utiliser la combinaison des 2 ou l'un des 2. Pour ne pas prendre en compte ces facteurs de dissipation, il faut qu'il soit mis à 1.
Il existe 2 modes possibles pour le fac_up: le mode martien (en fonction de l'altitude) et le mode vénusien (en fonction de la pression). Dans le 1er cas, il faut renseigner l'altitude où démarre la zone de transition et l'épaisseur de cette zone. Dans le 2ème cas, il faut renseigner la pression où démarre la zone de transition et l'échelle de hauteur de cette zone de transition.
Il est vivement conseillé de tracer votre profil de dissipation à partir des équations (dans vert_prof_dissip_icosa_lmdz.f90) afin d'être certain de leur allure.
E) Rayleigh Friction
Spiga et al 2020: This drag plays the role devoted to surface friction on terrestrial planets, which allows to close the angular momentum budget through downward control (Haynes and McIntyre, 1987; Haynes et al., 1991). This could also be regarded as a zeroth-order parameterization for Magneto-HydroDynamic (MHD) drag as a result of Lorenz forces acting on jet streams putatively extending to the depths of Saturn's interior (Liu et al., 2008; Galanti et al., 2019), much deeper than the shallow GCM's model bottom.
Comme Liu and Schneider (2010), cette couche de frottement ne s'exerce pas aux régions équatoriales car à l’extérieur du cylindre tangeant du rayon équatorial, les cylindres convectifs ne coupent pas cette couche et ne subissent pas les frottements liés aux effets de la MHD.
Le paramètre rayleigh_limlat correspond à la latitude maximale de part et d'autre de l'équateur où cette friction n'est pas observée.
Pour le temps (rayleigh_friction_tau), il est similaire à celui utilisé par Liu and Schneider (2010). Elle est fixée à 100 jours terrestres. Mais attention, on fonction de la valeur, la dynamique peut évoluer dans votre simulation.
F) Conservation
Dans run_icosa.def:
check_conservation = detailed itau_check_conserv = 320
Le itau_check_conserv est comme itau_physics pour que la contribution de la physique à AAM ne soit pas nulle. de plus il est coûteux d'appeler les diagnostics trop souvent.
S. Lebonnois, C. Covey, A. Grossman, H. Parish, G. Schubert, R. Walterscheid, P. Lauritzen, and C. Jablonowski. Angular momentum budget in General Circulation Models of superrotating atmospheres: A critical diagnostic. Journal of Geophysical Research (Planets), 117:E12004, 2012.
P. H. Lauritzen, J. T. Bacmeister, T. Dubos, S. Lebonnois, and M. A. Taylor. Held-Suarez simulations with the Community Atmosphere Model Spectral Element (CAM-SE) dynamical core: A global axial angular momentum analysis using Eulerian and floating Lagrangian vertical coordinates. Journal of Advances in Modeling Earth Systems, 6:129-140, 2014.
G) Traceur
- Branche master version du 04/04/2022 dans le dossier Jupiter (traceur)
La version actuelle des fichiers de réglage dans le dossier Jupiter est réglée pour utiliser des traceurs. Si vous souhaiter utiliser le modèle sans traceur, il vous faut modifier les fichiers suivants :
run_icosa.def
nqtot = 0
traceur.def
0
context_dynamico.xml (ligne 125)
<field id="q_start" name="q" grid_ref="grid_q_start" prec="8"/>
H) Bottom du modèle
- refaire tourner le 1D
- changer la pression
psurfdans rcm1d.def - changer
ichoice=1et changertref(ex: 10b: 330K)
- changer la pression
- refaire tourner le 3D (avec makestart) en changeant
preffdans[nom_de_la_planète]_const.def
4. Physical parameterizations
A) Cycle diurne
diurnal = .false. Car le temps radiatif est beaucoup plus long sur les planètes géantes.
B) Anneaux
[Pour saturne uniquement] Il est possible d'ajouter l'ombre des anneaux dans le callphys.def:
rings_shadow = .true.
C) Collision-induced absorption data
L'absorption induite par collision est importante dans le transfet radiatif sur les planètes géantes, notamment celle provenant de H2. Cependant, le rapport ortho-para du H2 est différent selon les planètes et peut modifier fortement le chauffage sur ces planètes. Pour le cas des géantes gazeuses, le rapport ortho-para est normal (rapport 3:1). Mais pour le cas des géantes glacées, le rapport ortho-para est à l'équilibre. Par défaut, le rapport ortho-para est normal (H2orthopara_mixture = normal). Pour les géantes glacées, il faut mettre H2orthopara_mixture = equilibrium .
Si vous voulez modifier le fichier utilisé dans un cas de CIA, il faut aller dans le code (dynamico-giant/code/LMDZ.GENERIC/libf/phystd/interpolate????.F90) et changer le nom du fichier (assurez-vous que ce fichier soit dans votre répertoire DATAGENERIC/continuum_data).
Dans la version actuelle, il n'existe pas d'option qui permet de ne pas utiliser certaines contributions de CIA. Par exemple, si votre atmosphère est composé de H2, He et CH4 et que vous ne voulez pas des contributions venant du CH4, il vous faut commenter dans le modèle ces contributions.
Les fichiers k-corrélées doivent se situer dans votre répertoire DATAGENERIC/corrk_data .
E) Cpp mode
cpp_mugaz_mode=0 pour que la valeur de cpp et de mugaz proviennent de la dynamique. cpp_mugaz_mode=1 pour forcer la valeur de cpp et de mugaz dans le callphys.def (à utiliser dans le makestart uniquement) cpp_mugaz_mode=2 pour calculer automatiquement à partir de données de références à 300 K et du gases.def (à éviter)
F) Generic n-layer aerosols (replaces the former 2-layer and NH3 cloud)
Ce mode permet de créer des couches d'aérosols/nuages ayant une opacité (aeronlay_tauref) à une longueur d'onde donnée (aeronlay_lamref), un rayon de particule fixe (aeronlay_size) situés à une pression fixe et en fonction des propriétés optiques des particules (optprop_aeronlay_vis et optprop_aeronlay_ir). Pour le cas de l'altitude, on peut choisir soit (aeronlay_choice = 1) une pression max (aeronlay_ptop) et une pression min (aeronlay_pbot), soit (aeronlay_choice = 2) une pression min (aeronlay_pbot) et une échelle de hauteur (aeronlay_sclhght). On peut également choisir la variance effective pour les rayons de particules.
Le nombre de couche de nuage (nlayero) est égal au nombre de scatterers dans la compilation (option -s ).
Les fichiers de propriétés optiques doivent être dans votre DATAGENERIC.
Un exemple pour 3 couches:
aeronlay = .true. nlayaero = 3 aeronlay_tauref = 2.5 0.04 0.1 aeronlay_lamref = 0.8e-6 0.8e-6 0.16e-6 aeronlay_choice = 2 2 1 aeronlay_pbot = 1.5e5 1.6e5 20. aeronlay_ptop = 1.1e5 1. 1. aeronlay_sclhght = 0.1 2.0 1 aeronlay_size = 0.5e-6 0.05e-6 0.5e-6 aeronlay_nueff = 0.3 0.3 0.3 optprop_aeronlay_vis = optprop_aerosol2_vis.dat optprop_aerosol3_vis.dat optprop_carbon4_vis.dat optprop_aeronlay_ir = optprop_aerosol2_ir.dat optprop_aerosol3_ir.dat optprop_carbon4_ir.dat
G) Panaches thermiques
- Comme pour toutes nouvelles simulations, il faut commencer par un run 1D de plusieurs décennies permettant d'obtenir un profil de température (
temp_profile.txt) et les coefficients ap et bp (apbp.txt) en équilibre radiatif-convectif pour la planète que l'on étudie. Ce run 1D s'effectue dans les dossiers jupiter1d, saturn1d, neptune1d et/ou uranus1d, avec ces options pour le callphys.def :
callrad = true calladj = true tous les autres mots clés à false (y compris calltherm).
Faire un "makestart" run : permet d'obtenir les fichiers restart à partir du profil de température initial. Il s'agit du run dont l'état initial est le profil de température créé par run 1D (
temp_profile.txtest appliquer à chaque point de grille horizontale du modèle). L'état initial est ainsi une planète isotherme horizontalement mais qui varie verticalement. Pour ce run, aucun traceur ne va être utilisé dans le modèle. Néanmoins, il faut renseigner au modèle le nombre de traceurs que nous souhaitons utiliser avec le schéma des panaches thermiques afin qu'il puisse créer la dimension nq et le champ q dans les fichiersrestart_icosa.ncetrestartfi.nc. dans callphys.def :traceur = true
dans run_icosa.def :
nqtot = 2(par exemple, h2o_vap et h2o_ice) dans traceur.def :2 h2o_vap h2o_iceAjout des quantités pour chaque traceur. Ici, nous ajoutons les profils pour chacun des traceurs dans les fichiers
restart_icosa.ncetrestartfi.nc"à la main" en utilisant le programme python/processing_codes/tracer_settings.pypour obtenir des fichiers restart avec la bonne abondance d'eau dans le cas présent.Commencer la simulation : Il ne reste plus qu'à lancer la simulation 3D avec les nouveaux fichiers restart et les réglages suivant : dans callphys.def :
traceur = true calltherm = true # thermal plume model options: divmpl = true r_aspect_thermals = 2.0 tau_thermals = 0.0 betalpha = 0.9 afact = 0.7 fact_epsilon = 2.e-4 alpha_max = 0.7 fomass_max = 0.5 pres_limit = 2.e5 water = true watercond = true waterain = true evap_prec = true
dans run_icosa.def : nqtot = 2 dans traceur.def :
2
h2o_vap
h2o_ice
Enfin, ajouter la déclaration et l'écriture des variables relatives à l'utilisation du schéma des thermiques (h2o_vap, h2o_ice, w_plm) dans les fichiers XML de la physique.
H) Rotation
Dans [name_of_planet]_const.def, changer le taux de rotation.
Cela n'a jamais été testé avec 0, mais cela pourrait créer des problèmes (ex: beta). vérifier que omega dans saturn_const.def n'intervient pas dans la physique LMDZ.GENERIC/libf/phystd/
I) Appel à la physique
Il faut changer à deux endroits en réglant la même valeur: - dans run_icosa.def, changer itau_physics ; - dans run.def, changer iphysiq. Ces paramètres sont exprimés en pas de temps dynamique. Pour appeler la physique à chaque pas de temps dynamique, régler ces paramètres à 1.
Attention: Lorsque l'on change dans le run_icosa.def la valeur de itau_physics, faire bien attention à la valeur du champ itau_adv (qui controle la frequence d'advection des traceurs par la dynamique, compté en pas de temps dynamique). Il faut que itau_physics soit un multiple de itau_adv. Cela veut dire, que l'on advecte notre champ de traceurs plus souvent (ou autant de fois) que l'on appelle la physique, qui peut transformer ces traceurs.
5. Ecriture des fichiers
A) Nombre d'écriture dans chaque Xhistins.nc
Prenons l'exemple de l'écriture d'un fichier sur une simu Uranus pour 1000j
Nombre d'écriture = day_step X ndays/(itau_physics X output_freq)
- ndays = 1000
- day_step = 200
- itau_physics = 50
- output_freq = 200
On a 20 sorties tous les 1000 jours.
Globalement, il est conseillé d'avoir 800 à 1000 sorties minimum par année planétaire (voir beaucoup plus en fonction de votre étude).
B) Ajouter une variable
Pour ajouter une variable dans le fichier de sortie Xhistins.nc, il faut l'ajouter dans le field_group correspondant et s'assurer que cette variable est présente en tant que variable de sortie dans dynamico-giant/code/LMDZ.GENERIC/libf/phystd/physiq_mod.F90. Si ce n'est pas le cas, il faut faire un call writediagfi de la variable puis l'ajouter dans un call send_xios_field.
C) Forcer XIOS à écrire tous les ....
Il faut utiliser l'attribut (ici par ex pour forcer l'écriture tous les ts (==time step) ; d'autres délais doivent être possible): sync_freq="1ts" dans le <file id=... .... > concerné.
D) XIOS server ou client
- XIOS client
use_server=FalseBroadwell 24 processeurs sur 28 - XIOS server
use_server=trueBroadwell 24 processurs sur 28 + 4 processeurs pour XIOS
Le cas 1 est 3 min plus lent que le cas 2 -- sur 2h20...(!) parce qu'on fait peu de sorties.
6. Running the model
A) Compilation
Pour compiler le modèle, il suffit d'aller dans le dossier avec vos fichiers .def et .xml et d'utiliser le script compile_occigen.sh si on est sur occigen ou compile_ciclad.sh si on est sur ciclad
./compile_occigen.sh
Les options qui peuvent être modifiées/ajoutées sont - -s : le nombre de scatterers - -parallel: mpi ou mpi_omp - -arch: le nom du ficher d'architecture - -arch_path: le chemin vers ce fichier - -job: 8 (conseillé) - -full: compile le code entièrement - -debug: pour trouver un éventuel bug. A ENLEVER obligatoirement s'il n'y a plus de bug car multiplie par 5 votre temp de calcul.
En 1D, d'autres options sont nécessaires: - -t: nombre de traceurs - -d: nombre de niveaux verticaux - -parallel: none (en 1D uniquement)
- Versions fonctionnelles de dynamico-giant
Jupiter - XIOS revision 1583
- ICOSAGCM revision 756
- IOIPSL revision 339
- FCM_V1.2 revision 12
- Physics revision 2228 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
but check Physics version 2142 and 2180
Saturne (référence Spiga 2020, à vérifier) - DYNAMICO --> Revision: 756 - PHYSICS --> Revision: 2005 - XIOS --> Revision: 1583 - IOIPSL --> Revision: 310?
Saturne (simulation de référence sur 61 niveaux, Bardet et al. Icarus 2021) - XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 310
- FCM_V1.2 revision 12
- Physics revision 2005 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Saturne (simulation avec la GWD paramétrisation sur 61 niveaux, chapitre 6 PhD Bardet) - XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 310
- FCM_V1.2 revision 12
- Physics revision 2213 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Saturne (simulation sur 96 niveaux, Bardet et al. Nature Astronomy 2022) - XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 431
- FCM_V1.2 revision 12
- Physics revision 2305 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Saturne (simulation avec la GWD paramétrisation sur 96 niveaux, chapitre 6 PhD Bardet) - XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 431
- FCM_V1.2 revision 12
- Physics revision 2403 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Uranus & Neptune (old version) - XIOS revision 1944 - ICOSAGCM revision 765 - IOIPSL revision 431 - FCM_V1.2 revision 12
- Physics revision 2413 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Uranus & Neptune (new version) - XIOS revision 2203 - ICOSAGCM revision (20/08/2021) - IOIPSL revision 450 - FCM_V1.2 revision 12
- Physics revision 2555 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Jupiter, Saturne, Uranus & Neptune [OCCIGEN VERSION] - XIOS revision 2319 - ICOSAGCM revision (90f7138a60ebd3644fbbc42bc9dfa22923386385) - IOIPSL revision 453 - FCM_V1.2 revision 12
- Physics revision 2655 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Jupiter, Saturne, Uranus & Neptune [IRENE VERSION] - XIOS revision 2399 - ICOSAGCM revision (4fbd393a9051fd9c1a5b662683f6ad8d0dc2867c) - IOIPSL revision 6234 - FCM_V1.2 revision 12
- Physics revision 2842 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
B) Run 1D
Pour une simulation 1D, 1 seul CPU suffit.
To run on Occigen:
sbatch job_mpi
To run on Irene:
ccc_msub job_mpi
To see evolution of your job on Occigen:
squeue -u username
To see evolution of your job on Irene:
ccc_mpp -u username
To kill your job on Occigen:
scancel id_of_your_job
To kill your job on Irene:
ccc_mdel id_of_your_job
Une fois que votre simulation est fini, lancer le script python printapbp.py qui vous permettra d'obtenir les fichiers apbp.txt et temp_profile.txt nécessaires pour les simulations en 3D.
C) Run 3D
Pour lancer une première simulation, il faut tout d'abord le faire une seule fois dans le dossier makestart de l'un des dossiers jupiter, saturne, uranus ou neptune. Ce dernier permettra de créer les premiers fichiers restart_icosa.nc et restartfi.nc. Ensuite, il suffit revenir dans le dossier parent (jupiter, saturne, uranus, neptune) et de lancer ses simulations.
- MPI
Avant de lancer une simulation, il est nécessaire de déterminer - le nombre de procs - le nombre de noeuds
Il faut avant tout savoir qu'il y a 10 x nsplit_i x nsplit_j domaines. Ces domaines sont divisés en plusieurs cellules. Il y a (nbp)²/(nsplit_i x nsplit_j) cellules par domaine. Ce nombre est égal au nombre de MPI process.
Il est important de ne pas faire des tuiles "trop petites" et donc de garder nsplit_i et nsplit_j tels que nbp/nsplit_{i,j} >=10-15 (plutôt 15). Car il y a des calculs "redondants" faits sur les bords du domaine. Admettons que ton domaine soit 10 x 10 (100 cellules par domaine). Le nombre de cellules au bord du domaine est égal à 2 x nsplit_i + 2 x nsplit_j - 4. Dans ce cas, le nombre de cellules au bord est de 36. Cela signifie que le bord du domaine correspond à 36% du domaine (36 points sur 100). Si le domaine est 15 x 15, alors le bord du domaine n'est plus que de 24.8 %. On aimerait avoir des domaines les plus grands possibles mais il faut aussi voir que chaque cellule correspond à une colonne à résoudre... Et c'est là ou il faut expérimenter un peu pour trouver l'optimum entre le nombre total de proc à employer et le gain effectif (temps total "facturé" au vu du nombre de procs monopolisés pour une simu donnée). Ehouarn
Il faut au total que le nombre de procs soit égal à 10 x nsplit_i x nsplit_j qu'il faut répartir entre les noeuds.
Pour les noeuds, il faut tenir compte qu'un noeud sur Occigen, c'est 24 (ou 28) procs. Disons 24. Quand on demande N procs, le système te donne M noeuds, soit M X 24 procs (les noeuds ne sont pas partagés avec d'autres applications). Et bien sûr on te facturera ces M noeuds, même si tu n'utilises pas tous les procs. Donc il faut s'efforcer de tomber juste et demander un multiple de 24 procs. Même raisonement si tu demandes à utiliser des noeuds de 28 procs.
Par exemple, nsplit_i = 4 et nsplit_j = 6. 4 x 6 est égal à 24. On utilisera donc 10 noeuds pour avoir 240 nprocs.
Prenons un autre exemple. nsplit_i = 5 et nsplit_j = 6. 5x6 = 30 Or il y a 24 procs par noeuds. Il faudra utiliser 13 noeuds. Et ce ne sont pas 300 procs qui seront facturés au total mais 312.
Mais comme vu plus haut, en fonction de la configuration de tes domaines, il est possible que le 2nd exemple coûte moins cher en heure CPU que le 1er exemple car il sera plus rapide.
- OpenMP / MPI
Dans le cas mixte OpenMP/MPI, il est nécessaire de renseigner le nombre de tâche OpenMP et de MPI threads. Dans le script pour le job, on a :
cpus-per-task et OMP_NUM_THREADS correspondent au nombre de tâche OpenMP ntask-per-node correspond au nombre de tâche par noeud
Pour déterminer la configuration à utiliser en OpenMP/MPI, il faut respecter ces règles: - ntask-per-node x nodes correspond au nombre total de MPI threads qu'il devrait y avoir (c'est-à-dire dans le cas MPI seul). - cpus-per-task x ntask-per-node doit être égale ou inférieur au nombre de procs par noeuds (24 ou 28- sur Occigen) - cpus-per-task devrait être ~égal au nombre de niveaux verticaux / 10 - OMP_NUM_THREADS doit être égal au cpus-per-task - omp_level_size (dans le fichier run_icosa.def) devrait être ~égal au nombre de cpus-per-task. (dans le cas MPI seul, il faut le laisser égal à 1)
Au total, le nombre total de nprocs est égal à cpus-per-task x ntask-per-node x nodes
En reprenant le premier exemple MPI (on suppose qu'on a 40 niveaux verticaux), on aura - cpus-per-task = 4 - ntask-per-norde = 6 On a bien cpus-per-task x ntask-per-norde <= 24. Le nombre de noeuds doit être égal à 40 car 6x40 = 240 nprocs MPI Le nombre de procs qu'on utilisera au total sera de 960.
- Comment déterminer le nombre d'heure CPU (consommation en heure)?
Heures CPU = durée_de_la_simu X nombre de procs total facturé
Si on veut connaître le nombre d'heure CPU qu'on consommera à partir d'un test, voici le calcul
Heures CPU = durée_de_la_simu_test X nombre de procs total facturé X nombre de jour qu'on veut simulé / (nombre de jour simulées pendant la simu test)
D) Optimisation
Pour savoir quelle est la meilleure configuration en MPI seul, en OpenMP/MPI et entre les 2, des tests ont été effectués. Ces tests ont été réalisés sur Uranus à nbp80 sur les noeuds Broadwell (28 procs par noeud). 1000 jours uraniens ont été simulés à chaque fois.
La comparaison du MPI seul et du mixte OpenMP/MPI permet de tirer la conclusion que le mixte OpenMP/MPI est beaucoup plus rapide (facteur 2.5 à 3) mais consomme plus (facteur 1.1 à 1.7).
E) Visualizing the output files
- Comment comparer deux fichiers? file1.nc file2.nc
ncdiff file1.nc file2.nc output.nc
- Comment coller plusieurs fichiers file1.nc file2.nc file3.nc
ncrcat file1.nc file2.nc file3.nc output.nc
Avec juste la vitesse u et v: ncrcat -v u -v v file1.nc file2.nc file3.nc output.nc
Avec tous les fichiers fileXXX.nc disponibles ncrcat file*.nc output.nc
- Comment tracer les champs et divers diagnostics en moyenne zonale?
- mettre à jour planetoplot et planets
- utiliser precast.py dans planetoplot/examples/ppclass_additional/dynanalysis
changer les options au début (il y a des exemples), exemple pour Saturne
fileAP="Xhistins_42.nc" p_upper,p_lower,nlev = 4.0e2,2.5e5,40 targetp1d = np.logspace(np.log10(p_lower),np.log10(p_upper),nlev) myp = planets.Saturn day_per_year = 24430. short = False includels = False charx = "0,360" ispressure = False vartemp = "temperature" outfile = "precast.nc" nopole = True
- Il faut que apbp.txt soit présent !
Le résultat se trouve dans le fichier indiqué dans outfile.
F) Debug
- Pour debugger
Réglez info_level à 100 dans le fichier iodef.xml .
- Erreur en début de run, SEGMENTATION FAULT dans la routine advect.f90 (ICOSAGCM/ppsrc/transport) au niveau de l'appel à cross_product2
La routine cross_product2 effectue des produits vectoriels. Cette erreur vient du fait qu'en essayant de faire des optimisations de calcul, le compilateur se prend "les pieds dans le tapis" et ne sait plus faire le produit vectoriel. Pour empêcher le compilateur de faire des optimisations trop poussées, il faut ajouter l'option -fp-model strict au niveau du mot-clé %BASE_FFLAGS dans tous les fichiers .fcm du modèle (LMDZ.COMMON / ICOSAGCM / ICOSA_LMDZ).
G) Pas de radiatif et perturbations
- faire descendre le modèle
- modifier le code pour perturbations vitesse et pas température
- compiler (voir README.md)
changer dans callphys.def (dans saturn/makestart)
surfalbedo = 1.0 surfemis = 0.0
changer dans callphys.def (dans saturn et saturn/makestart)
corrk = .false. enertest = .true. randompert = 1
- voir si on garde le flux interne ou non (
intheat) - refaire les états initiaux (dans makestart)
faire un run court pour voir sur les spectres l'injection d'Ek
H) PROFILING with DYNAMICO-Giant
-pg: Generate extra code to write profile information suitable for analysis program gprof. You must use this option when compiling the sources files you want data about and you must also use it when linking. 1. For DYNAMICO-Giant: in the arch.fcm file of each part of the model (ICOSAGCM, ICOSA_LMDZ, LMDZ.COMMON and XIOS), you have to add the option -pg to %PROD_FFLAGS and %BASE_LD 2. Compile as usual 3. Execute your code as usual 4. Run gprof tool:
gprof icosa_lmdz.exe goon.out > analysis.txt
OLD WIKI (DEPRECATED, JUST FOR REFERENCE/INFORMATION)
``Welcome to the dynamico-giant wiki!
pour debugger
régler info_level à 100 dans le fichier iodef.xml
pourquoi diurnal=.false.
le temps radiatif est beaucoup plus long sur les géantes
comment changer la fréquence d'appel à la physique?
il faut changer à deux endroits en réglant la même valeur - dans run_icosa.def, changer itau_physics - dans run.def, changer iphysiq ces paramètres sont exprimés en pas de temps dynamique. pour appeler la physique à chaque pas de temps dynamique, régler ces paramètres à 1
comment changer la résolution
comment choisir le nombre de processeurs pour la résolution
voir commentaires sur run_icosa.def
########################################### ## There must be less MPIxOpenMP processes than the 10 x nsplit_i x nsplit_j tiles ## typically for pure MPI runs, let nproc = 10 x nsplit_i x nsplit_j ## it is better to have nbp/split >~ 10 ########################################### #### 40 noeuds de 24 processeurs = 960 procs nsplit_i=12 nsplit_j=8
Ehouarn: Une règle à suivre est de ne pas faire des tuiles "trop petites" et donc de garder nbsplit_i et nbsplit_j tels que nbp/nbsplit >=10-15 (plutôt 15). Car il y a des calculs "redondants" faits sur les bords du domaine. Or si ton domaine est 1010, le bord du domaine c'est 38% du domaine (38 points sur le bord pour 100 points en tout) alors que si ton domaine est 1515, le bord du domaine n'est plus que 56/(1515) = 25.7%. 56=215+213 (pour ne pas compter 2 fois les coins). On voudrait du coup des domaine les plus grand possible, clairement, mais il faut aussi voir que chaque domaine c'est nbpnbp colonnes à résoudre sur ce même proc... Et c'est là ou il faut expérimenter un peu pour trouver l'optimum entre le nombre total de proc à employer et le gain effectif (temps total "facturé" au vu du nombre de procs monopolisés pour une simu donnée).
_Pour les noeuds, il faut tenir compte qu'un noeud sur Occigen, c'est 24 (ou 28) procs. Disons 24. Quand on demande N procs, le système te donne M noeuds, soit M*24 procs (les noeuds ne sont pas partagés avec d'autres applications). Et bien sûr on te facturera ces M noeuds, même si tu n'utilises pas tous les procs. Donc il faut s'efforcer de tomber juste et demander un multiple de 24 procs. Même raisonement si tu demandes à utiliser des noeuds de 28 procs._
comment changer la rotation?
dans saturn_const.def, changer le taux de rotation jamais testé avec 0, mais pourrait créer des problèmes (ex: beta) vérifier que omega dans saturn_const.def n'intervient pas dans la physique LMDZ.GENERIC/libf/phystd/
la dissipation c'est où?
laplacien itéré: hyperviscosity - tau_graddiv,tau_gradrot,tau_divgrad: periode (plus c'est petit, plus la dissipation est efficace) - nitergdiv,nitergrot,niterdivgrad: ordre du laplacien (plus c'est grand, plus on dissipe sélectivement les petites échelles)
comment changer la grille cible du remapping on-the-fly
dans context_lmdz_physics.xml changer les paramètres ni_glo et nj_glo par exemple pour remapper sur du lat/lon à 360 pts en latitude et 720 pts en longitude (0.5°) <domain id="dom_regular" ni_glo="720" nj_glo="360" type="rectilinear">
Forcer XIOS à écrire tous les ....
Il faut utiliser l'attribut (ici par ex pour forcer l'écriture tous les ts (==time step) ; d'autres délais doivent être possible): sync_freq="1ts" dans le <file id=... .... > concerné.
comment changer le bottom du modele
- refaire tourner le 1D
- changer la pression
psurfdans rcm1d.def - changer
ichoice=1et changertref(ex: 10b: 330K)
- changer la pression
- refaire tourner le 3D (avec makestart) en changeant
preffdanssaturn_const.def
comment changer la fréquence de sortie des Xhistins.nc
les commandes XIOS sont appelées depuis la physique (dans la config présente) dans context_lmdz_physics.xml il suffit de changer output_freq attention ts se comprend comme le pas de temps physique (voir donc dans run_icosa.def les paramètres dt et itau_physics pour le connaître) par exemple, pour des sorties tous les 20 jours Saturne
<file id="histins"
name="Xhistins"
output_freq="40ts"
type="one_file"
enabled=".true.">
pas de radiatif et perturbations
- faire descendre le modèle
- modifier le code pour perturbations vitesse et pas température
- compiler (voir README.md)
changer dans callphys.def (dans saturn/makestart)
surfalbedo = 1.0 surfemis = 0.0
changer dans callphys.def (dans saturn et saturn/makestart)
corrk = .false. enertest = .true. randompert = 1
- voir si on garde le flux interne ou non (
intheat) - refaire les états initiaux (dans makestart)
faire un run court pour voir sur les spectres l'injection d'Ek
comment comparer deux fichiers? file1.nc file2.nc
ncdiff file1.nc file2.nc output.nc
comment coller plusieurs fichiers file1.nc file2.nc file3.nc
ncrcat file1.nc file2.nc file3.nc output.nc avec juste la vitesse u et v ncrcat -v u -v v file1.nc file2.nc file3.nc output.nc avec tous les fichiers fileXXX.nc disponibles ncrcat file*.nc output.nc
comment tracer les champs et divers diagnostics en moyenne zonale?
- mettre à jour planetoplot et planets
- utiliser precast.py dans planetoplot/examples/ppclass_additional/dynanalysis
changer les options au début (il y a des exemples), exemple pour Saturne
fileAP="Xhistins_42.nc" p_upper,p_lower,nlev = 4.0e2,2.5e5,40 targetp1d = np.logspace(np.log10(p_lower),np.log10(p_upper),nlev) myp = planets.Saturn day_per_year = 24430. short = False includels = False charx = "0,360" ispressure = False vartemp = "temperature" outfile = "precast.nc" nopole = True
- Il faut que apbp.txt soit présent !
le résultat se trouve dans le fichier indiqué dans outfile
bilan de moment cinétique
dans run_icosa.def
check_conservation = detailed itau_check_conserv = 160
le itau_check_conserv est comme itau_physics pour que la contribution de la physique à AAM ne soit pas nulle. de plus il est coûteux d'appeler les diagnostics trop souvent.
S. Lebonnois, C. Covey, A. Grossman, H. Parish, G. Schubert, R. Walterscheid, P. Lauritzen, and C. Jablonowski. Angular momentum budget in General Circulation Models of superrotating atmospheres: A critical diagnostic. Journal of Geophysical Research (Planets), 117:E12004, 2012.
P. H. Lauritzen, J. T. Bacmeister, T. Dubos, S. Lebonnois, and M. A. Taylor. Held-Suarez simulations with the Community Atmosphere Model Spectral Element (CAM-SE) dynamical core: A global axial angular momentum analysis using Eulerian and floating Lagrangian vertical coordinates. Journal of Advances in Modeling Earth Systems, 6:129-140, 2014.
XIOS server ou client
- XIOS client
use_server=FalseBroadwell 24 processeurs sur 28 - XIOS server
use_server=trueBroadwell 24 processurs sur 28 + 4 processeurs pour XIOS
cas 1 est 3 min plus lent que cas 2 -- sur 2h20...! parce qu'on fait peu de sorties
Optimisation
- 40 noeuds au lieu de 50 noeuds, même temps de calcul...! Testé dans la branche https://github.com/aymeric-spiga/dynamico-giant/tree/work_61levels
-
sync_freq=40ts(ou multiple de, si 40ts est la fréquence d'écriture) dans context_lmdz_physics.xml doit être inclus sinon il n'est pas réglé et XIOS transfère tout à la fin du run ce qui prend du temps (et peut occasionner des problèmes de mémoire)
Versions fonctionnelles de dynamico-giant
Jupiter
- XIOS revision 1583
- ICOSAGCM revision 756
- IOIPSL revision 339
- FCM_V1.2 revision 12
- Physics revision 2228 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
but check Physics version 2142 and 2180
Saturne (référence Spiga 2020, à vérifier)
- DYNAMICO --> Revision: 756
- PHYSICS --> Revision: 2005
- XIOS --> Revision: 1583
- IOIPSL --> Revision: 310?
Saturne (simulation de référence sur 61 niveaux, Bardet et al. Icarus 2021)
- XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 310
- FCM_V1.2 revision 12
- Physics revision 2005 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Saturne (simulation avec la GWD paramétrisation sur 61 niveaux, chapitre 6 PhD Bardet)
- XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 310
- FCM_V1.2 revision 12
- Physics revision 2213 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Saturne (simulation sur 96 niveaux, Bardet et al. Nature Astronomy 2022)
- XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 431
- FCM_V1.2 revision 12
- Physics revision 2305 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Saturne (simulation avec la GWD paramétrisation sur 96 niveaux, chapitre 6 PhD Bardet)
- XIOS revision 1583
- ICOSAGCM revision 765
- IOIPSL revision 431
- FCM_V1.2 revision 12
- Physics revision 2403 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Uranus & Neptune (old version)
- XIOS revision 1944
- ICOSAGCM revision 765
- IOIPSL revision 431
- FCM_V1.2 revision 12
- Physics revision 2413 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
Uranus & Neptune (new version)
- XIOS revision 2203
- ICOSAGCM revision (20/08/2021)
- IOIPSL revision 450
- FCM_V1.2 revision 12
- Physics revision 2555 (LMDZ.COMMON, LMDZ.GENERIC, ICOSA_LMDZ, ARCH)
works on 03/03/2022 for all HEAD
Les fichiers d'architecture (Installation sur un nouveau cluster)
En pratique LMDZ.COMMON, ICOSA_LMDZ et IOIPSL peuvent utiliser exactement le même fichier arch.fcm ; mais celui pour ICOSAGCM est légèrement différent (les %FPP_DEF diffèrent, peut-être aussi le %FPP).
make_icosa_lmdz doit être lancé avec -arch_path ../ARCH puisque les arch.env et arch.path communs se trouvent dans ../ARCH (l'option -arch_path ne concerne d'ailleurs que les fichiers arch.env et arch.path; le fichier arch.fcm recherché sera toujours celui dans le "arch" de chacun des modèles. Donc il faut bien mettre pour chacun des quatre modèles (LMDZ.COMMON, ICOSA_LMDZ,IOIPSL et ICOSAGCM) le arch.fcm dans le sous-dossier arch/ du modèle correspondant.
XIOS est écrit en C++, et pas en Fortran. Le fichier arch.fcm correspondant est donc nécessairement différent de celui des quatre autres modèles. Pour créér ce arch.fcm, prendre exemple sur les fichiers .fcm déjà présent dans XIOS/arch/, avec une architecture similaire à celle du nouveau cluster. En particulier, il faut utiliser des versions de gcc/gfortran > 6+. Il faut absolument avoir une bibliothèque HDF5 compilée en parallèle, ainsi que netcdf-C et netcdf-fortran (et peut être aussi netcdf-Cxx) compilé en parallèle. Sans ça, il sera peut-être possible de compiler le modèle, mais pas de lancer une simulation en utilisant XIOS.
Commencer une nouvelle simulation en utilisant le schéma des panaches thermiques
- Comme pour toutes nouvelles simulations, il faut commencer par un run 1D de plusieurs décennies permettant d'obtenir un profil de température (
temp_profile.txt) et les coefficients ap et bp (apbp.txt) en équilibre radiatif-convectif pour la planète que l'on étudie. Ce run 1D s'effectue dans les dossiers jupiter1d, saturn1d, neptune1d et/ou uranus1d, avec ces options pour le callphys.def :
callrad = true calladj = true tous les autres mots clés à false (y compris calltherm).
Faire un "makestart" run : permet d'obtenir les fichiers restart à partir du profil de température initial. Il s'agit du run dont l'état initial est le profil de température créé par run 1D (
temp_profile.txtest appliquer à chaque point de grille horizontale du modèle). L'état initial est ainsi une planète isotherme horizontalement mais qui varie verticalement. Pour ce run, aucun traceur ne va être utilisé dans le modèle. Néanmoins, il faut renseigner au modèle le nombre de traceurs que nous souhaitons utiliser avec le schéma des panaches thermiques afin qu'il puisse créer la dimension nq et le champ q dans les fichiersrestart_icosa.ncetrestartfi.nc. dans callphys.def :traceur = true
dans run_icosa.def :
nqtot = 2(par exemple, h2o_vap et h2o_ice) dans traceur.def :2 h2o_vap h2o_iceAjout des quantités pour chaque traceur. Ici, nous ajoutons les profils pour chacun des traceurs dans les fichiers
restart_icosa.ncetrestartfi.nc"à la main" en utilisant le programme python/processing_codes/tracer_settings.pypour obtenir des fichiers restart avec la bonne abondance d'eau dans le cas présent.Commencer la simulation : Il ne reste plus qu'à lancer la simulation 3D avec les nouveaux fichiers restart et les réglages suivant : dans callphys.def :
traceur = true calltherm = true # thermal plume model options: divmpl = true r_aspect_thermals = 2.0 tau_thermals = 0.0 betalpha = 0.9 afact = 0.7 fact_epsilon = 2.e-4 alpha_max = 0.7 fomass_max = 0.5 pres_limit = 2.e5 water = true watercond = true waterain = true evap_prec = true
dans run_icosa.def : nqtot = 2 dans traceur.def :
2
h2o_vap
h2o_ice
Enfin, ajouter la déclaration et l'écriture des variables relatives à l'utilisation du schéma des thermiques (h2o_vap, h2o_ice, w_plm) dans les fichiers XML de la physique.
Erreur en début de run, SEGMENTATION FAULT dans la routine advect.f90 (ICOSAGCM/ppsrc/transport) au niveau de l'appel à cross_product2.
La routine cross_product2 effectue des produits vectoriels. Cette erreur vient du fait qu'en essayant de faire des optimisations de calcul, le compilateur se prend "les pieds dans le tapis" et ne sait plus faire le produit vectoriel. Pour empêcher le compilateur de faire des optimisations trop poussées, il faut ajouter l'option -fp-model strict au niveau du mot-clé %BASE_FFLAGS dans tous les fichiers .fcm du modèle (LMDZ.COMMON / ICOSAGCM / ICOSA_LMDZ)
Branche master version du 04/04/2022 dans le dossier Jupiter
La version actuelle des fichiers de réglage dans le dossier Jupiter sont réglés pour utiliser des traceurs. Si vous souhaiter utiliser le modèle sans traceur, il vous faut modifier les fichiers suivants :
run_icosa.def
nqtot = 0
traceur.def
0
context_dynamico.xml (ligne 125)
<field id="q_start" name="q" grid_ref="grid_q_start" prec="8"/>
PROFILING with DYNAMICO-Giant:
-pg: Generate extra code to write profile information suitable for analysis program gprof. You must use this option when compiling the sources files you want data about and you must also use it when linking. 1. For DYNAMICO-Giant: in the arch.fcm file of each part of the model (ICOSAGCM, ICOSA_LMDZ, LMDZ.COMMON and XIOS), you have to add the option -pg to %PROD_FFLAGS and %BASE_LD 2. Compile as usual 3. Execute your code as usual 4. Run gprof tool:
gprof icosa_lmdz.exe goon.out > analysis.txt